COVID-19 na zdjęciach rentgenowskich wygląda podobnie do zapalenia płuc wywołanego innymi niż SARS-CoV-2 wirusami, a także zapalenia płuc pochodzenia bakteryjnego. Jednocześnie zdjęcie rentgenowskie powstaje szybciej niż wynik badania RT-PCR, będącego standardową metodą wykrywania wirusa SARS-CoV-2. Jest także łatwiejsze w wykonaniu w porównaniu do zdjęcia z tomografu komputerowego, m.in. dzięki urządzeniom przenośnym, które pozwalają na zrobienie zdjęcia pacjentowi w jego łóżku.
O wykorzystaniu zdjęć rentgenowskich w diagnostyce COVID-19 powstało do tej pory wiele artykułów (część metod z zeszłego roku można przejrzeć na przykład w tym artykule przeglądowym). Temat porusza również niedawno zakończony konkurs na Kaggle. Celem konkursu była klasyfikacja zdjęć rentgenowskich oraz detekcja nieprawidłowości na obrazie.
Wpis ten zawiera analizę zbioru danych dostarczonych przez organizatorów konkursu, omówienie wybranych rozwiązań do klasyfikacji i detekcji oraz przedstawienie otrzymanych wyników.
Zbiór danych
Na zbiór danych składają się obrazy treningowe i testowe w formacie DICOM oraz pliki csv z etykietami i przykładowe submission. W zbiorze treningowym jest 6334 obrazów, a w zbiorze testowym 1263.
data/
├── sample_submission.csv
├── test
│ ├── 00a81e8f1051
│ │ └── bdc0bb04ae1e
│ │ └── ced40f593496.dcm
│ ├── 00be7de16711
│ │ └── bfef2920427a
│ │ └── cea591e99b8a.dcm
│ ├── 00c7a3928f0f
│ │ └── 7476c897257c
│ │ └── f6ba3df9a8be.dcm
│ ├── 00d63957bc3a
│ │ └── 07919a1b758c
│ │ └── dbae9b9b9500.dcm
│ ├── 0a50336ef885
│ │ ├── 54b08a9fa42a
│ │ │ └── a24bc9ab2ea8.dcm
│ │ └── fe3fd685ebd2
│ │ └── e134e2ffdfaf.dcm
│ ...
│
├── train
│ ├── 00a76543ed93
│ │ └── 4a223cccbe04
│ │ └── ad8d4a5ba8f0.dcm
│ ├── 00a87235ca36
│ │ └── b7a93187765f
│ │ └── 09cf9767a7bf.dcm
│ ├── 0a1a3dd9e738
│ │ └── 79de130ea278
│ │ └── 64a776818efe.dcm
│ ├── 0a256a4617e8
│ │ ├── 6441f142de3a
│ │ │ └── 84a8af741443.dcm
│ │ └── c11e1e2da2fd
│ │ └── 467f412db5c4.dcm
│ ├── 0a29d44f380b
│ │ └── 22d381f8ae8c
│ │ └── eb61e3bdb2a5.dcm
│ ...
│
├── train_image_level.csv
└── train_study_level.csv
Ogólna ścieżka do danego obrazu wygląda następująco:
study/series/image
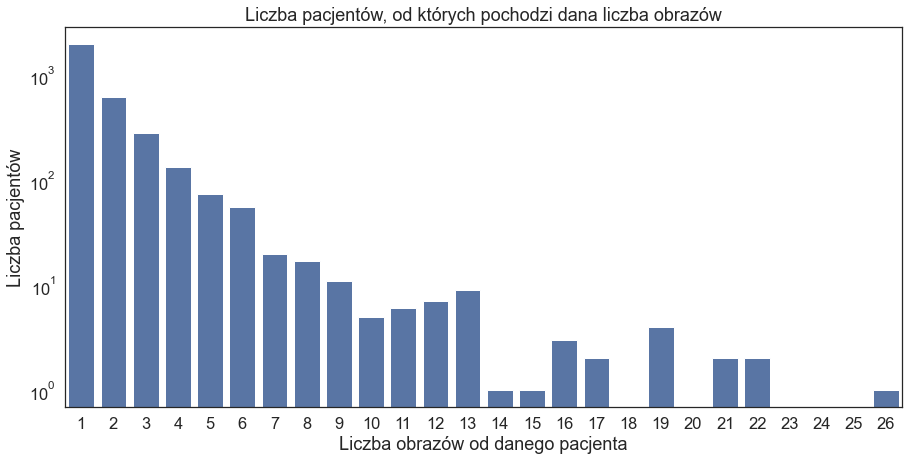
Każdemu study (badanie), series (seria) i image (obraz) odpowiada ID. W obrębie danego study możemy mieć więcej niż jeden obraz:
Analiza metadanych
Format DICOM oprócz samego obrazu zawiera również metadane dotyczące m.in. pacjenta (np. płeć, wiek), badania (np. data, badana część ciała), wykorzystanego urządzenia (np. modalność, producent). Przy udostępnianiu danych – na przykład na potrzeby konkursu Kaggle – część z tych danych jest usuwana/zamieniana na fikcyjne, aby chronić prywatność pacjenta. Przeanalizujmy niektóre z tych danych.
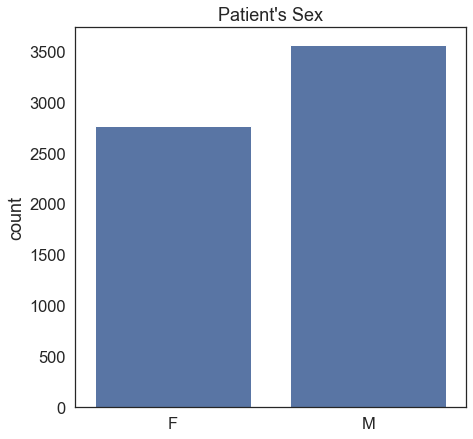
W zbiorze mamy nieco więcej mężczyzn (M – Male) niż kobiet (F – female), a ściślej mówiąc obrazów ogólnie pochodzących od mężczyzn, gdyż możemy mieć więcej niż jeden obraz od danego pacjenta:
Obrazy w zbiorze pochodzą z dwóch różnych modalności: CR (Computed Radiography) i DX (Digital Radiography, również skracane jako DR):
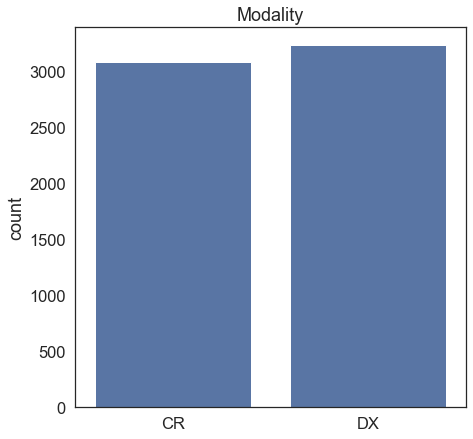
Różnica między nimi leży w sposobie rejestracji wiązki rentgenowskiej: CR wykorzystuje płyty luminoforowe (płyty IP), a DX cyfrowe matryce detekcyjne (Digital Detector Arrays, DDA). Obie techniki są technikami cyfrowymi w odróżnieniu od analogowej radiografii wykorzystującej klisze rentgenowskie. W razie wątpliwości, co oznacza dany skrót modalności w formacie DICOM, można zerknąć na przykład na tę tabelkę.
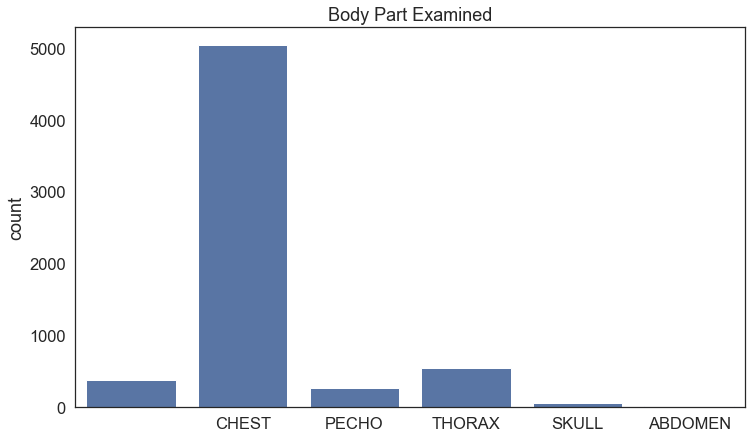
Istotną informacją jest, czy wszystkie obrazy w zbiorze przedstawiają interesującą nas część ciała, tj. klatkę piersiową:
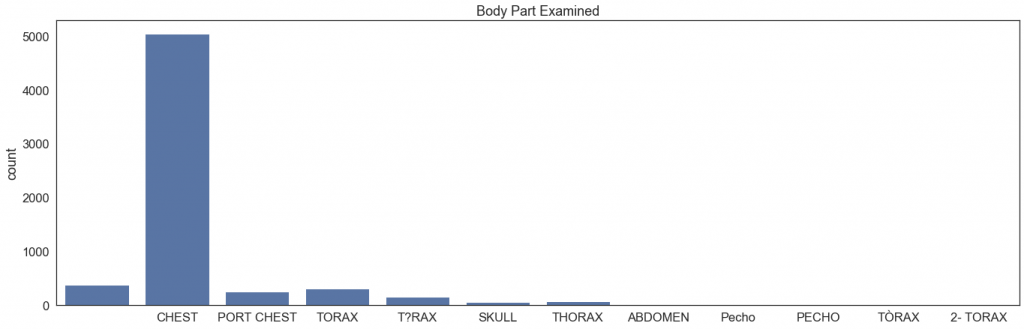
Widzimy, że w przypadku niektórych zdjęć pole 'Body Part Examined’ jest puste. Poza najliczniejszym 'CHEST’ trafiają się inne określenia na klatkę piersiową, takie jak 'THORAX’ oraz 'PECHO’ (po hiszpańsku) zapisane na kilka różnych sposobów. Znalazły się tutaj również inne części ciała (SKULL – czaszka, ABDOMEN – brzuch). Na szczęście nie ma ich zbyt dużo, więc można je przejrzeć manualnie – okazuje się, że są to błędy i obrazy przedstawiają jednak klatkę piersiową; w przeciwnym wypadku należałoby je wyrzucić ze zbioru, gdyż w konkursie interesują nas zmiany w płucach. Podobnie jest w przypadku obrazów bez wpisanej badanej części ciała, warto je przejrzeć i sprawdzić, czy faktycznie są to zdjęcia klatki piersiowej – okazuje się, że tak.
W celu łatwiejszej interpretacji możemy nieco skondensować wykres, łącząc ze sobą niektóre grupy i ujednolicając nazewnictwo (np. 'Pecho’ i 'PECHO’ utworzą razem jedną grupę 'PECHO’):
Obrazy w formacie DICOM posiadają informację, jak należy je interpretować, tj. jaka jest relacja pomiędzy wartością piksela a jego sposobem wyświetlania. Listę dostępnych wartości wraz z krótkim opisem można zobaczyć tu.
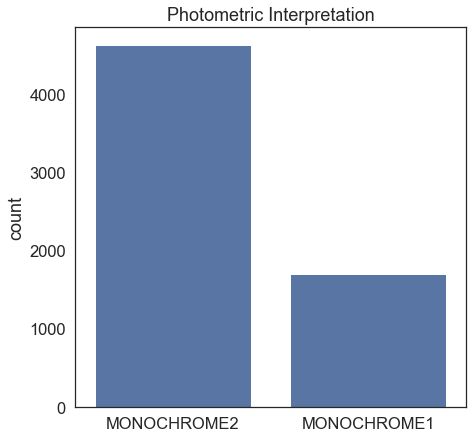
W naszym zbiorze mamy dwa rodzaje:
- MONOCHROME2 – małe wartości pikseli są ciemniejsze, a większe jaśniejsze (0 – czarny, 255 – biały),
- MONOCHROME1 – odwrotnie niż powyższe, małe wartości pikseli są jaśniejsze, a większe ciemniejsze (0 – biały, 255 – czarny).
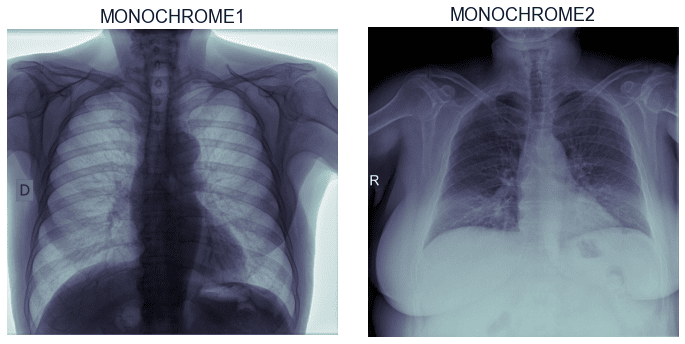
Gdy mamy w zbiorze więcej niż jeden rodzaj, przed trenowaniem sieci możemy zdjęcia przekonwertować do jednego rodzaju, ułatwiając w ten sposób zadanie modelowi. Ponieważ tutaj mamy więcej obrazów MONOCHROME2, obrazy MONOCHROME1 zostaną przekonwertowane na MONOCHROME2. W takim wypadku trzeba pamiętać o zamianie także przy przygotowywaniu zbioru testowego. Moglibyśmy również zostawić zdjęcia w ich oryginalnej reprezentacji, a taką konwersję wykorzystać jako data augmentation. Wytrenowana sieć mogłaby wtedy przyjmować na wejście obraz MONOCHROME1 lub MONOCHROME2.
Obrazy
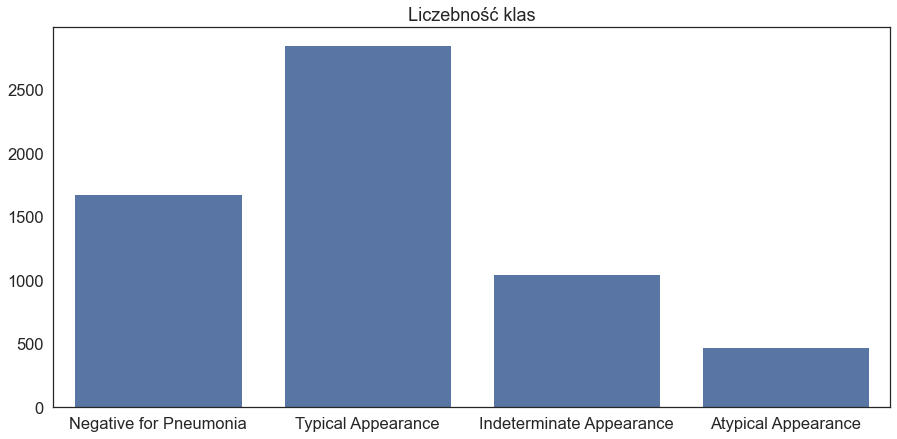
Obrazy w zbiorze należą do jednej z 4 klas:
- Negative for Pneumonia,
- Typical Appearance,
- Indeterminate Appearance,
- Atypical Appearance.
i mają różne rozdzielczości:
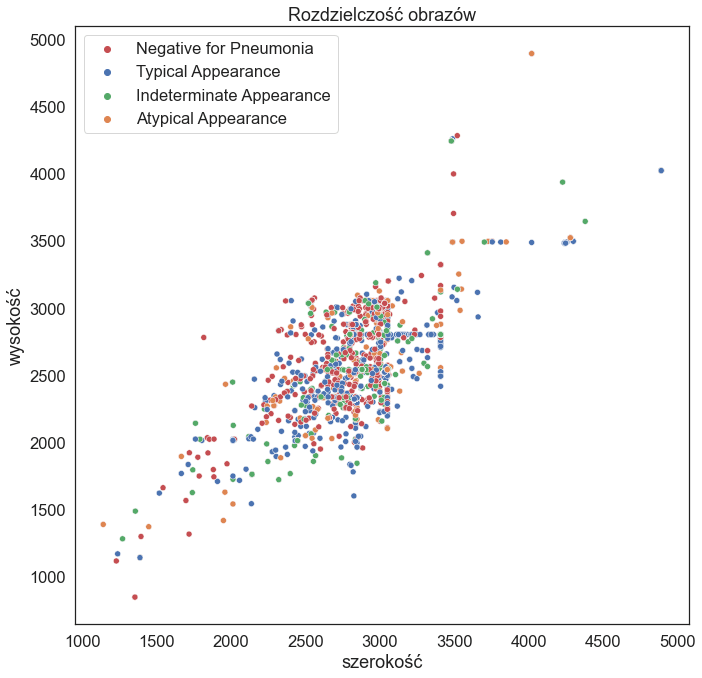
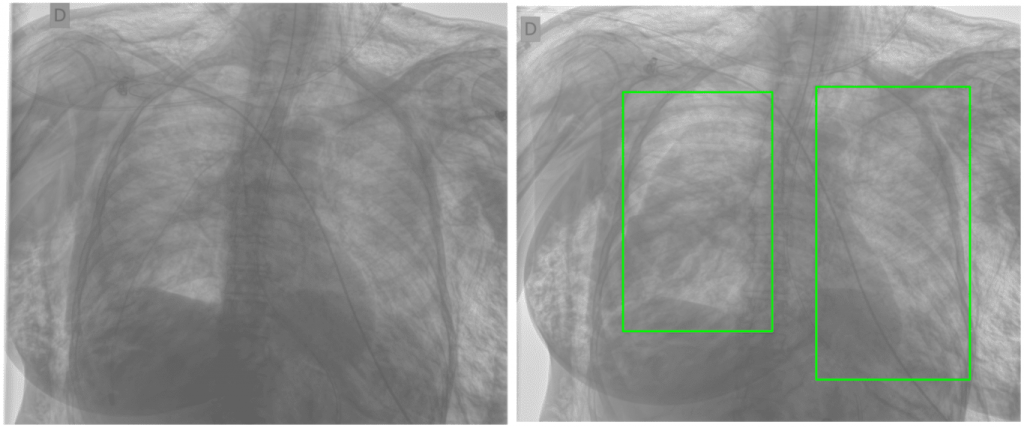
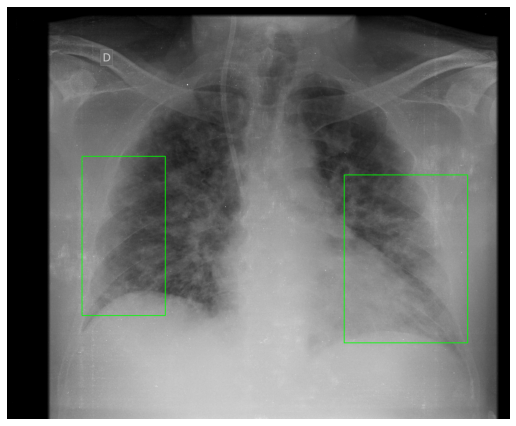
Opis klas można znaleźć w tabeli 1 w tym artykule. Widzimy, że zbiór jest niezbalansowany: najwięcej jest obrazów Typical Appearance, a najmniej Atypical Appearance. Należy wziąć to pod uwagę podczas eksperymentów i trenowania modeli. Spójrzmy jeszcze na przykładowe obrazy z każdej klasy wraz z narysowanymi bounding boxami otaczającymi zmiany patologiczne, które będziemy chcieli wykrywać:
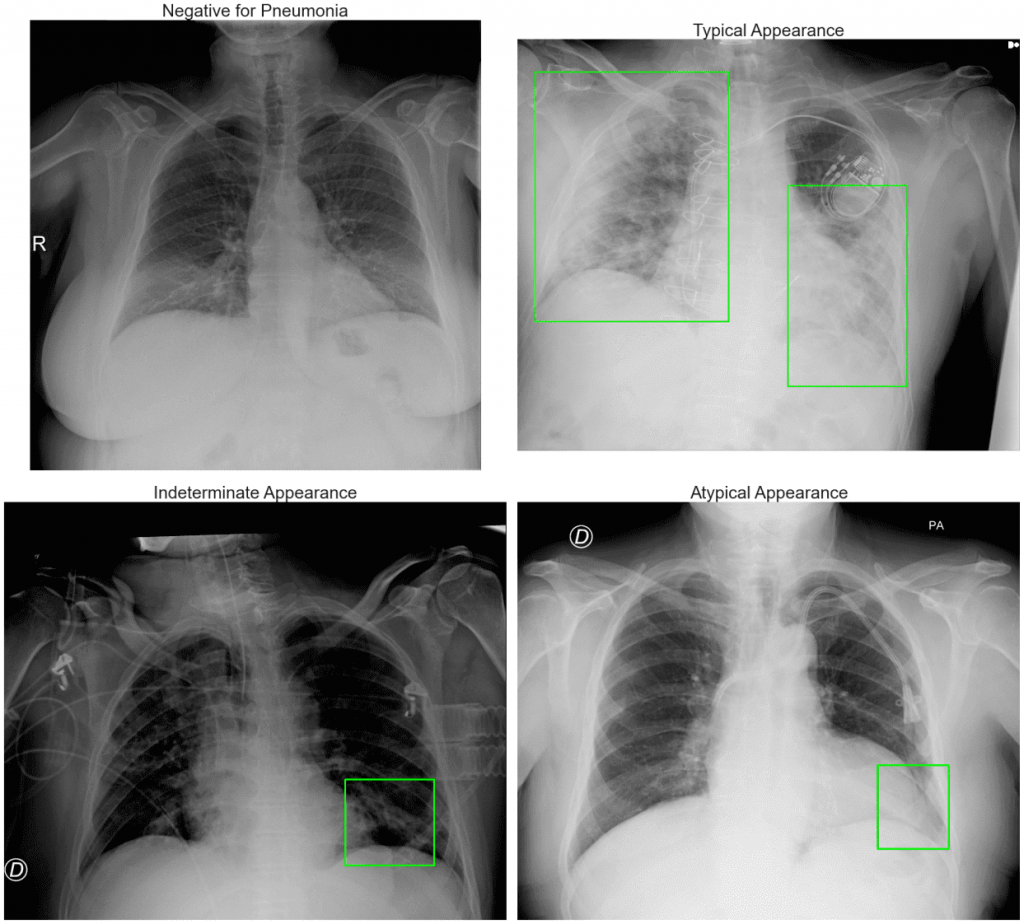
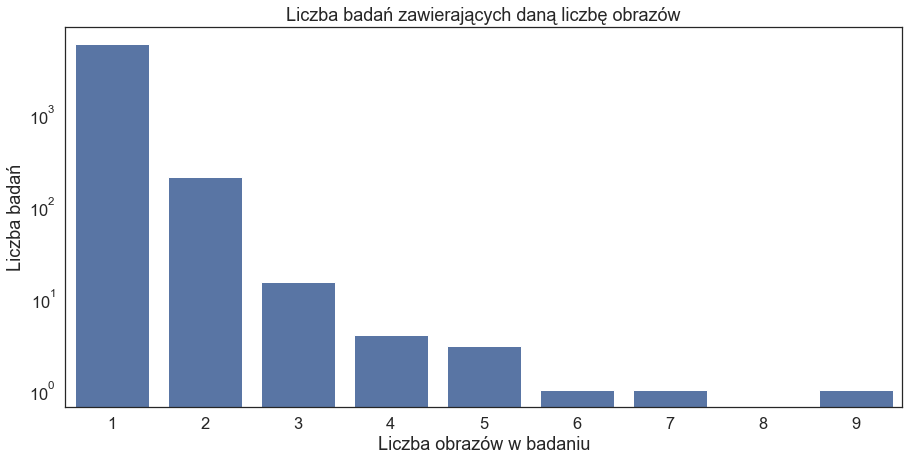
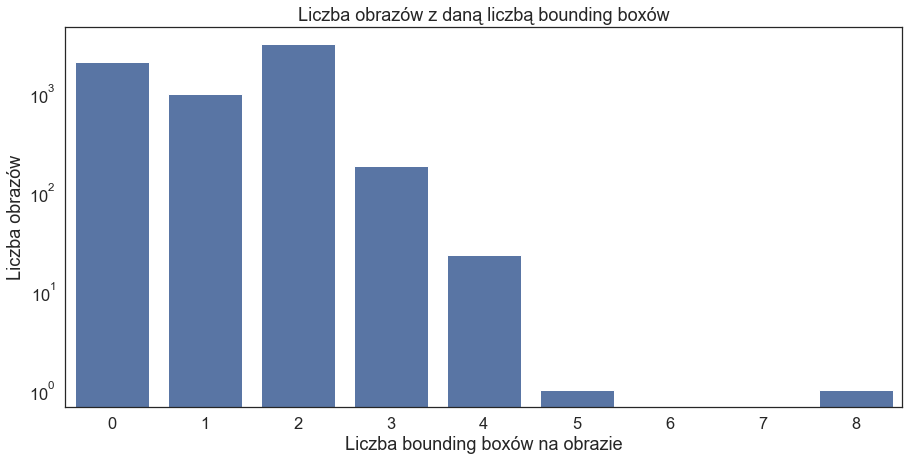
Liczba ramek na danym obrazie może być różna: od 0 (Negative for Pneumonia – brak zmian patologicznych; zaliczają się tu również duplikaty wspomniane poniżej) do aż 8 ramek.
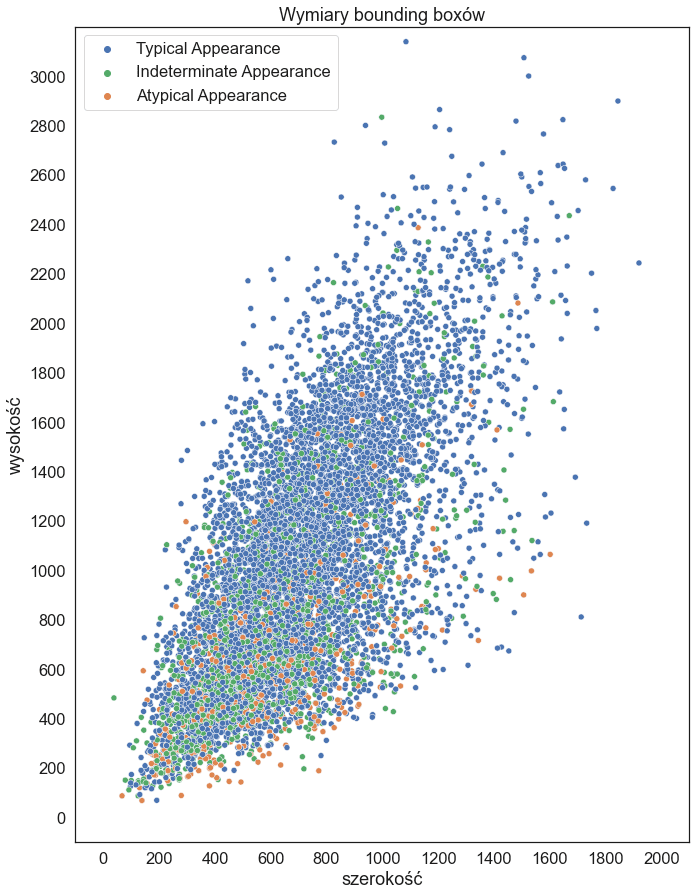
Wymiary bounding boxów są różne – niektóre są dość małe (jak np. dla Indeterminate Appearance powyżej), a niektóre duże, obejmujące całe płuco (jak np. dla Typical Appearance powyżej):
Duplikaty
W zbiorze treningowym trafiły się duplikaty. Nie są one jednak oczywiste na pierwszy rzut oka – nie chodzi o obrazy, które są identyczne i mają takie same metadane. Duplikaty znajdują się w obrębie jednego badania i są to prześwietlenia powtórzone przez technika lub różnie przetworzone (wyjaśnienie jednego z organizatorów konkursu). Tylko jeden z obrazów w takim badaniu ma zapisane bounding boxy; pozostałe obrazy bez ramek zgodnie z zaleceniem można usunąć ze zbioru treningowego.
Obrazy nietypowe
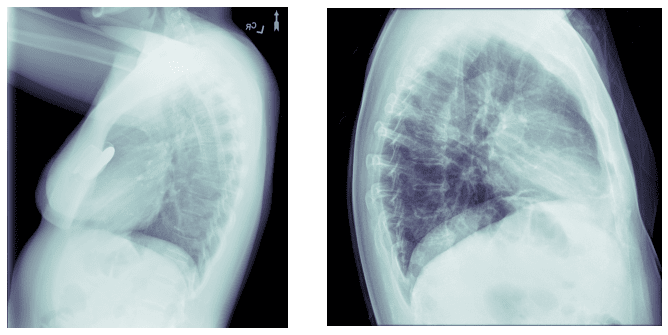
Poza duplikatami zdarzyły się w zbiorze obrazy nietypowe, które również nie było łatwo znaleźć od razu. Dwa z nich zostały usunięte ze zbioru treningowego – przedstawiały bowiem klatkę piersiową z boku:
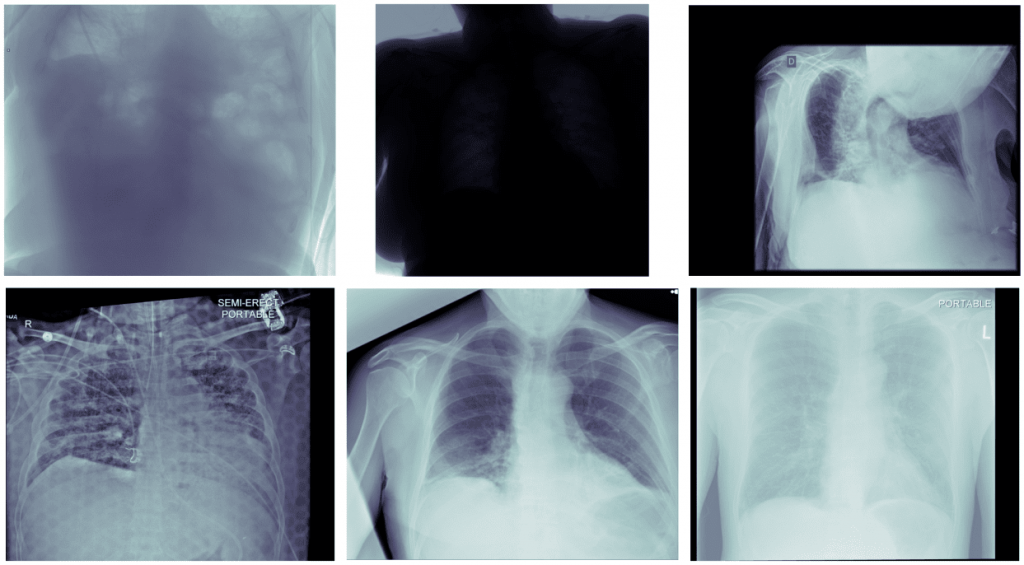
Do nietypowych obrazów można także zaliczyć obrazy bardzo jasne/ciemne, niewyraźne, z artefaktami, adnotacjami, z ledwo widocznymi płucami, itp. Te obrazy jednak zostały w zbiorze treningowym.
Eksperymenty
Zadanie składa się z dwóch części: klasyfikacji obrazu do jednej z 4 klas (na poziomie study) oraz detekcji zmian patologicznych w płucach (na poziomie image). Dla każdej predykcji podaje się:
- nazwę klasy (negative, typical, indeterminate, atypical) w przypadku study; opacity (cień, ogólnie zmiana w płucach) lub none (brak obiektów) w przypadku image,
- confidence – jak bardzo model jest pewny danej predykcji,
- współrzędne bounding boxa w formacie: xmin ymin xmax ymax – w przypadku study jest to zawsze '0 0 1 1′ odpowiadające ramce 1×1 piksel, dla image wpisujemy '0 0 1 1′, gdy nic nie zostało wykryte.
Przykładowe submission wygląda następująco:
id,PredictionString
00188a671292_study,negative 0.956145 0 0 1 1 typical 0.014483569 0 0 1 1 indeterminate 0.020749161 0 0 1 1 atypical 0.008622284 0 0 1 1
004bd59708be_study,negative 0.0024710842 0 0 1 1 typical 0.94615126 0 0 1 1 indeterminate 0.04259221 0 0 1 1 atypical 0.008785492 0 0 1 1
00508faccd39_study,negative 0.62312233 0 0 1 1 typical 0.24420053 0 0 1 1 indeterminate 0.11252862 0 0 1 1 atypical 0.02014848 0 0 1 1
...
66ca7bb52ab9_image,none 1 0 0 1 1
8ea54e20fd42_image,opacity 0.12 2670 1291 969 1159 opacity 0.12 2613 1261 1068 1210 opacity 0.11 2683 1476 944 1013
afc074e1bdb7_image,opacity 0.21 385 318 1018 1403 opacity 0.25 375 331 1048 1377 opacity 0.13 419 252 1137 1530 opacity 0.12 383 438 833 1308 opacity 0.13 356 429 886 1327 opacity 0.22 382 406 1013 1365 opacity 0.24 368 404 1049 1368
Powyższe submission przedstawia podejście – poruszane także w dyskusjach na Kaggle – które nieco poprawia wynik na tablicy wyników. Zostawiane są wszystkie wyniki, tj. dla study wpisuje się wszystkie klasy, nie tylko tę z największym confidence, a dla image wpisuje się wszystkie wykryte obiekty bez progowania – wszystkie które mają confidence > 0 (lub inny mały próg – powyżej było to akurat 0.1, ponieważ w moim przypadku obniżenie do 0.01 czasem pogarszało wynik).
Przygotowując submission do konkursu, otrzymane wyniki z obu części można potraktować oddzielnie lub rozpatrzyć je równolegle (na przykład jeśli detektor wykrył obiekty z małą pewnością, a klasyfikator uznał obraz za Negative for Pneumonia z dużą pewnością, to zostawiamy wynik klasyfikacji, a wynik detekcji zamieniamy na 'brak obiektów’).
Data Augmentation
Do data augmentation wykorzystane zostały następujące przekształcenia z biblioteki Albumentations:
transforms_ = [A.CLAHE(clip_limit=2.5),
A.HorizontalFlip(),
A.Rotate(15, border_mode=0, value=0),
A.MotionBlur([3,10]),
A.ShiftScaleRotate(rotate_limit=15, border_mode=0, value=0)]
if len(bboxes) >= 2:
transforms = transforms_ + [A.RandomSizedBBoxSafeCrop(img.shape[0], img.shape[1], p=0.5)]
RandomSizedBBoxSafeCrop – losowo przycinające obraz tak, aby bounding boxy nie zostały przycięte – było stosowane tylko dla obrazów, na których były co najmniej dwie ramki, aby nie stracić zbyt dużo obrazu, w szczególności płuc.
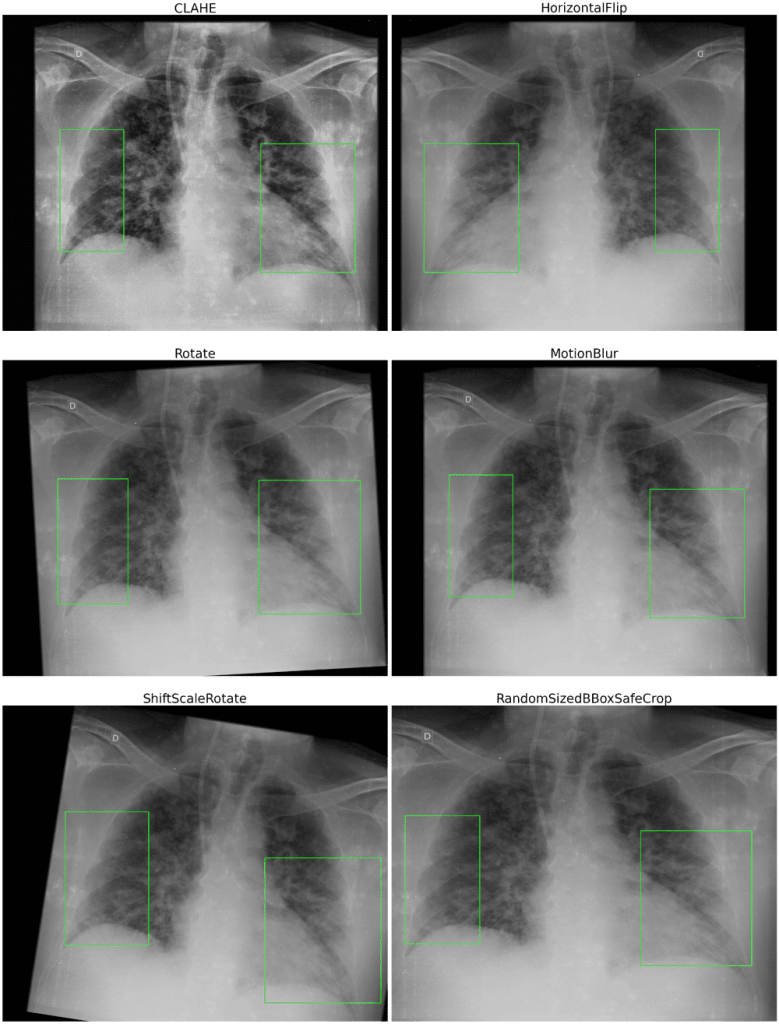
Data augmentation było stosowane zarówno w klasyfikacji, jak i detekcji. Dla klasyfikacji sprawdziłam również, czy data augmentation jedynie dla mało licznych klas poprawia wynik. Dodatkowo dla detekcji była używana mozaika:
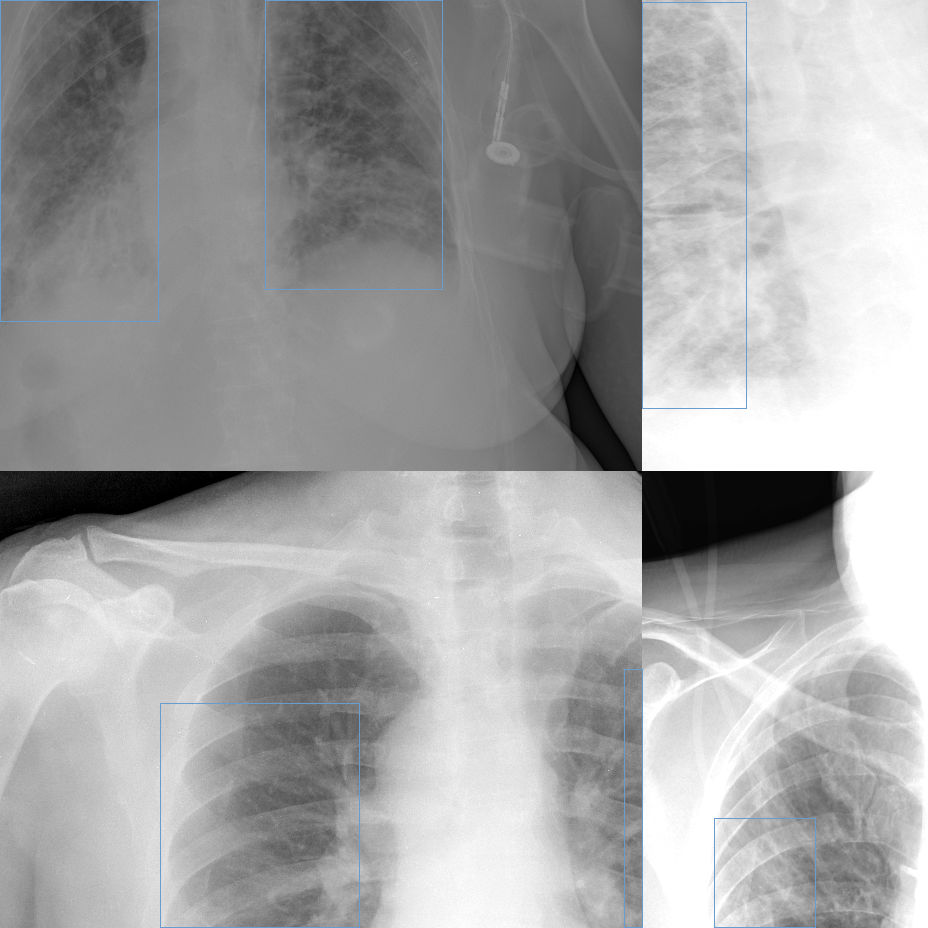
Klasyfikacja
Na framework do klasyfikacji wybrałam PyTorch Lightning, a jako klasyfikator EfficientNet V2 z repozytorium timm.
Jednym z problemów, jakie należało zaadresować, był brak balansu w danych, jak widzieliśmy na jednym z powyższych wykresów. Próby rozwiązania tego problemu były następujące:
- dodanie wag do funkcji kosztu,
- podniesienie wag do kwadratu,
- data augmentation tylko klas Indeterminate Appearance, Atypical Appearance (oznaczone jako dataaug2 w tabelce poniżej),
- zmiana podejścia i przejście na klasyfikację binarną oraz detekcję obiektów 3 klas – detektor wykrywa obiekty danej klasy na obrazie, tym samym informując o klasie, do której należy cały obraz.
Poniższa tabela przedstawia część eksperymentów. Wszystkie modele w tabeli miały rozmiar wejściowy 512×512 pikseli i efektywny batch size 64.
| kl. binarna | class weights | dataaug | dataaug2 | pretrained | AP | f1 | |
|---|---|---|---|---|---|---|---|
| 1 | ✓ | 0.437 | 0.614 | ||||
| 2 | ✓ | ✓ | 0.444 | 0.562 | |||
| 3 | ✓ | 0.400 | 0.515 | ||||
| 4 | ✓ | ✓ | ✓ | 0.447 | 0.551 | ||
| 5 | ✓ | ✓ | 0.445 | 0.560 | |||
| 6 | ✓ | ✓ | 0.395 | 0.537 | |||
| 7 | ✓ | ✓ | 0.764 | 0.766 | |||
| 8 | ✓ | ✓ | 0.802 | 0.774 |
Poza technikami wymienionymi w tabeli w trakcie eksperymentów sprawdziłam również inne:
- kontynuacja treningu z checkpointu z innymi parametrami,
- dobór learning rate,
- learning rate scheduler – StepLR,
- weight decay,
- więcej/mniej epok,
- większy rozmiar wejściowy,
- mniejszy batch.
Najlepsze modele wybrane do obliczenia ostatecznego wyniku w konkursie (można wybrać dwa submission) to model 5 i model 8.
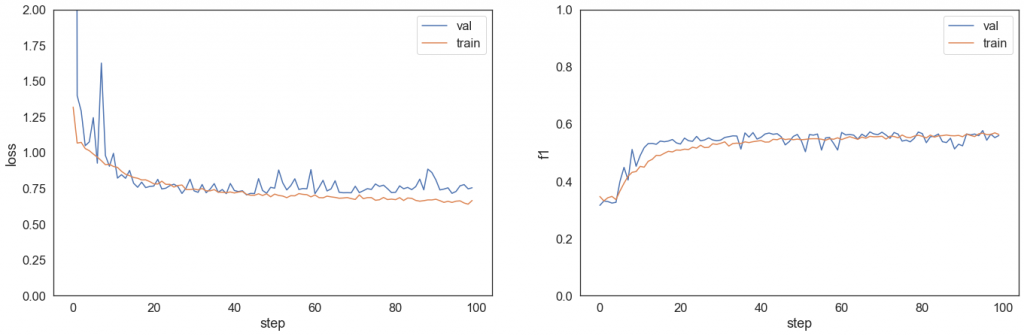
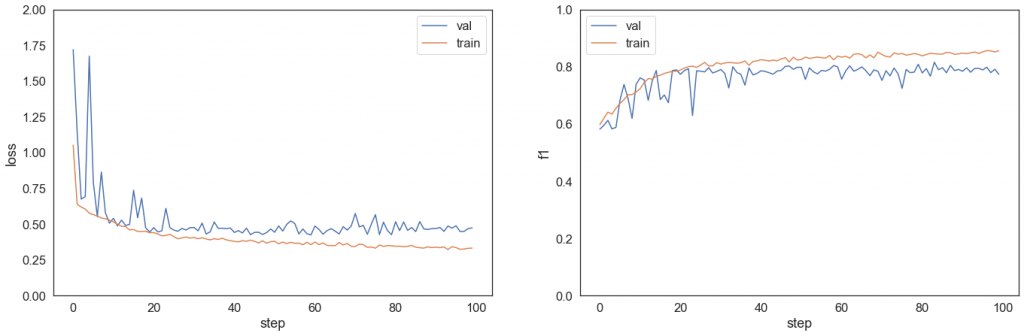
Przeanalizujmy macierze pomyłek oraz krzywe ROC i Precision-Recall wybranych modeli.
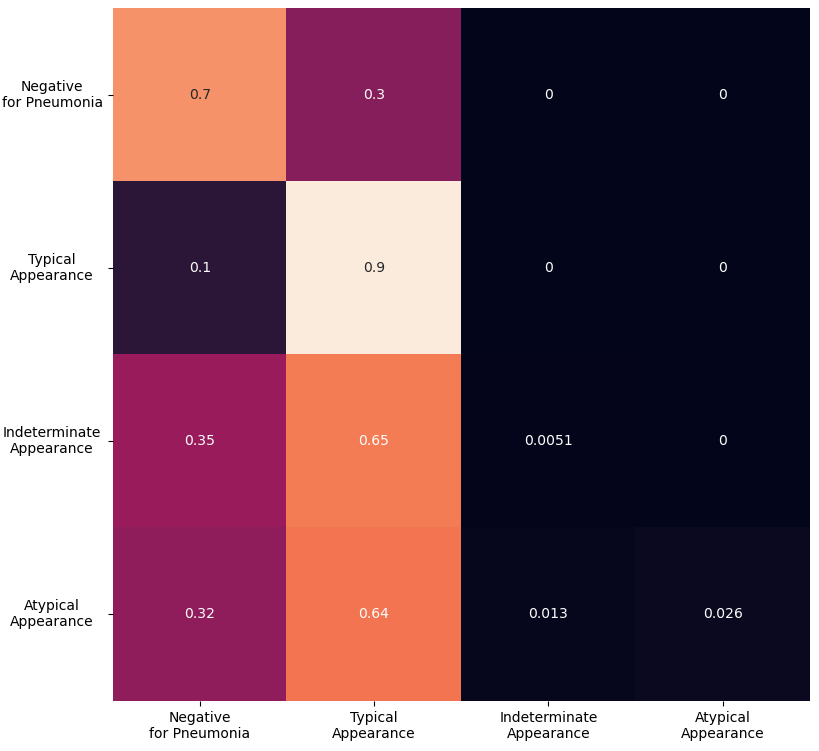
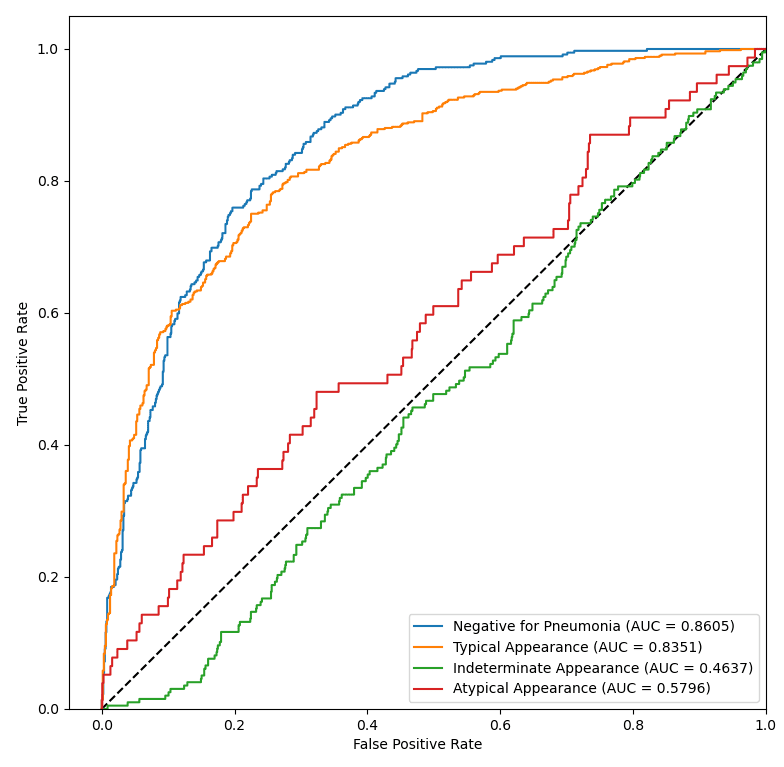
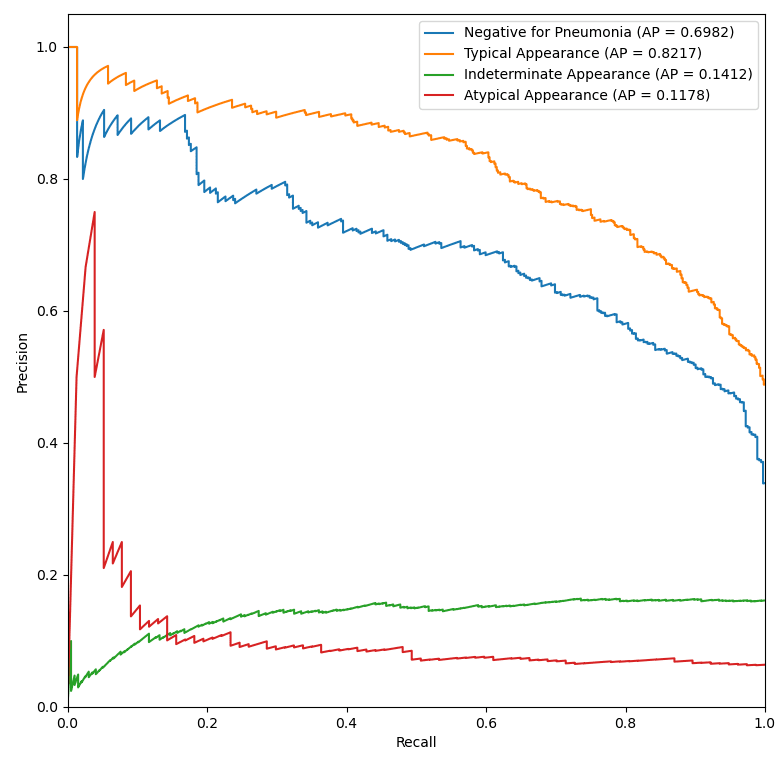
Niestety mimo wielu prób nie udało się osiągnąć obiecujących wyników dla wszystkich klas – obie niedoreprezentowane klasy są słabo rozpoznawane i klasyfikowane jako Negative for Pneumonia lub Typical Appearance. Widać to również na dwóch wykresach powyżej. Mało liczne klasy charakteryzują się niską średnią precyzją AP, a ich krzywe ROC leżą w pobliżu prostej x=y – klasyfikacja Atypical Appearance jest jedynie odrobinę lepsza od klasyfikacji losowej, a klasyfikacja Indeterminate Appearance jest wręcz gorsza niż klasyfikacja losowa (AUC < 0.5).
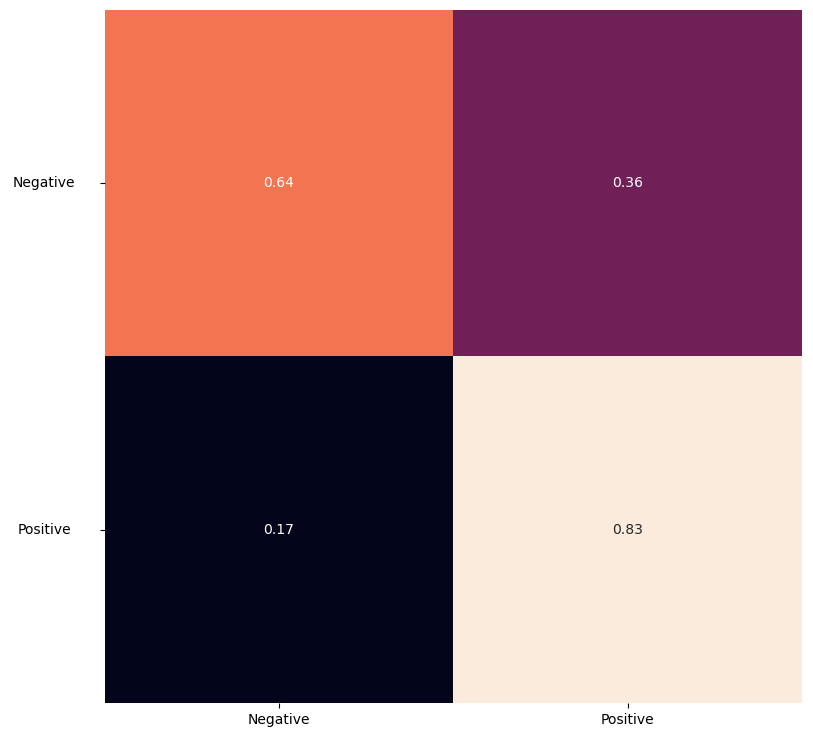
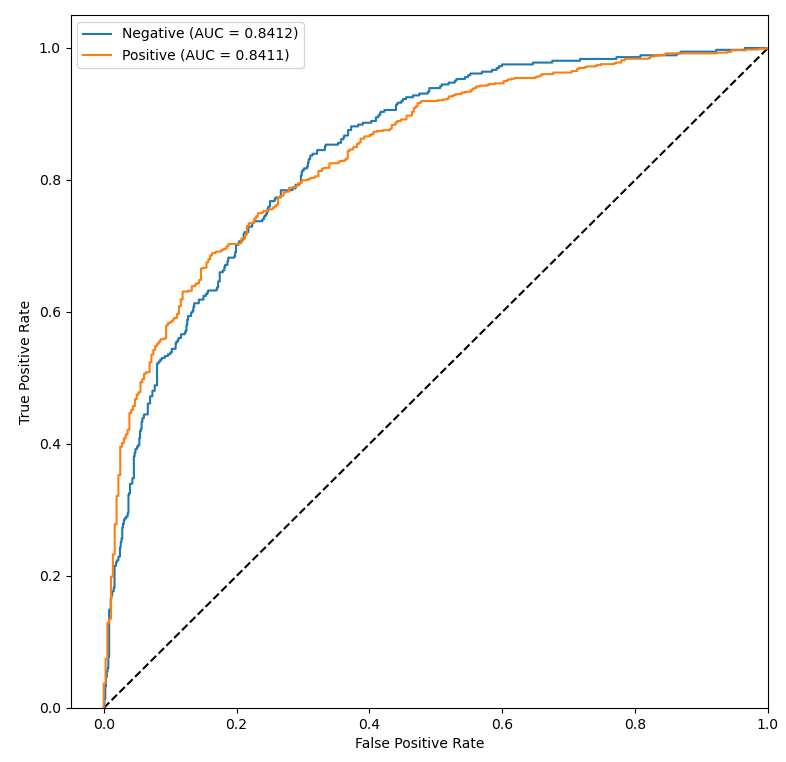
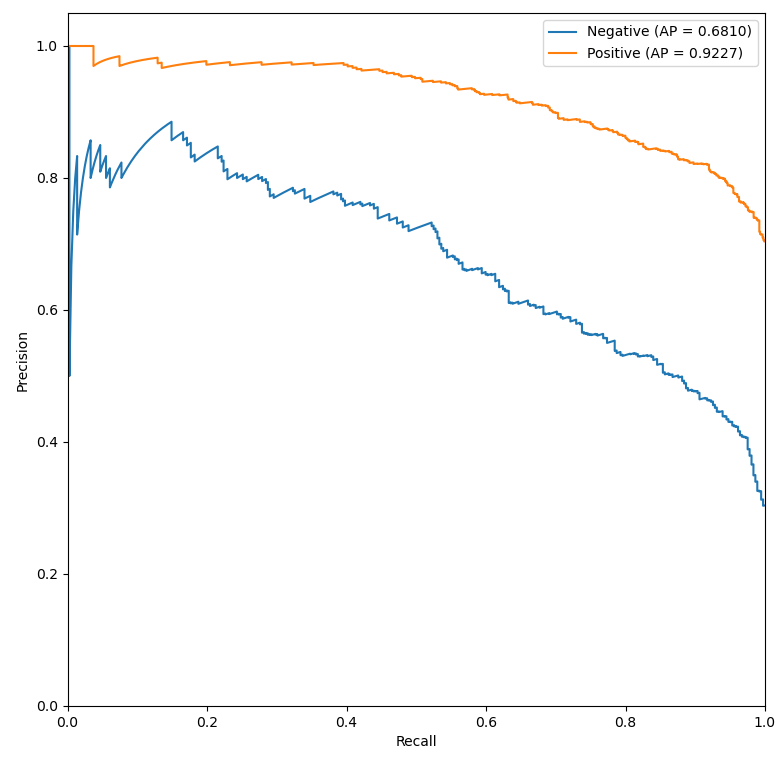
Wyniki klasyfikacji binarnej modelu 8 wyglądają bardziej zachęcająco, jednak ostatecznie submission z tym modelem osiągnęło niższy wynik na tablicy wyników. Jest to związane z tym, że takie podejście polega głównie na detektorze – detektor wykrywa zarówno docelową klasę, jak i obiekty, zatem detektor musiałby być bardziej efektywny.
Wyjaśnialność
Wyjaśnialność decyzji modelu jest w medycynie szczególnie istotna. Zobaczmy zatem, na jakiej podstawie modele klasyfikują obrazy do danej klasy. Do analizy klasyfikatorów wykorzystałam bibliotekę Captum. Wybrałam kilka metod:
- Saliency Maps – obliczenie gradientów względem obrazu wejściowego, poszczególne wynikowe wartości odpowiadają istotności pikseli o takiej samej lokalizacji na obrazie wejściowym,
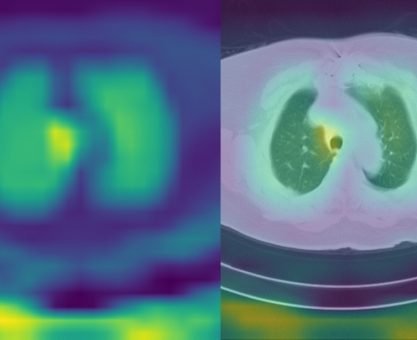
- GradCAM – obliczenie gradientów z wyjścia względem wybranej warstwy (tutaj ostatnia warstwa konwolucyjna), z grubsza zaznacza, które obszary są istotne przy predykcji danej klasy,
- Occlusion Sensitivity – zaznacza, jak bardzo dane obszary po zasłonięciu (np. czarnymi pikselami) zmieniają wynik dla danej klasy.
Razem z uzyskanymi heatmapami warto zwizualizować również bounding boxy – sprawdzimy, czy dla modelu istotne są obszary, które zaznaczyli lekarze jako zmiany patologiczne. Należy przy tym pamiętać, że w interpretacji modelu może również kryć się pewien haczyk i może ona być myląca – zobaczymy na przykład, które obszary są istotne, ale niekoniecznie będziemy wiedzieć, które konkretnie cechy danego obszaru wpłynęły na wynik.
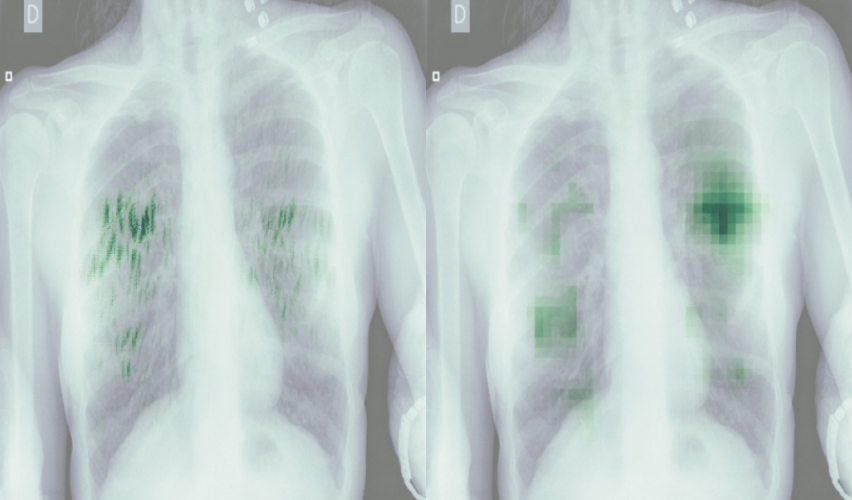
Spójrzmy na heatmapy dla modelu 5 uzyskane względem klasy prawdziwej. Dla poniższych obrazów model prawidłowo przewidział daną klasę.
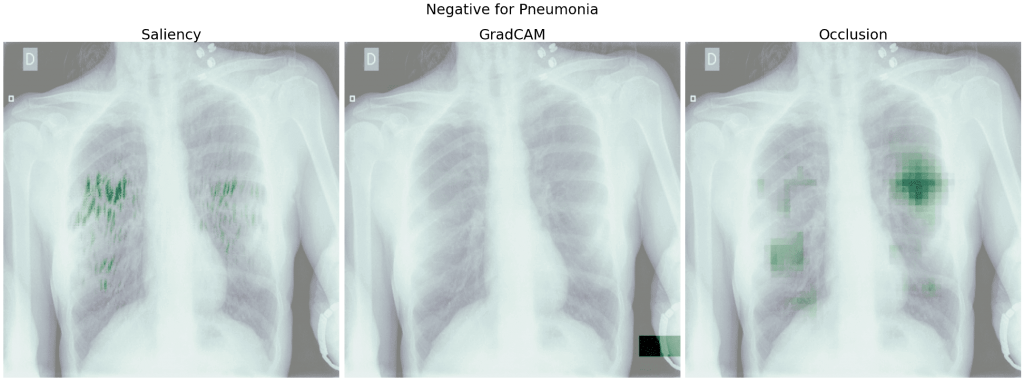
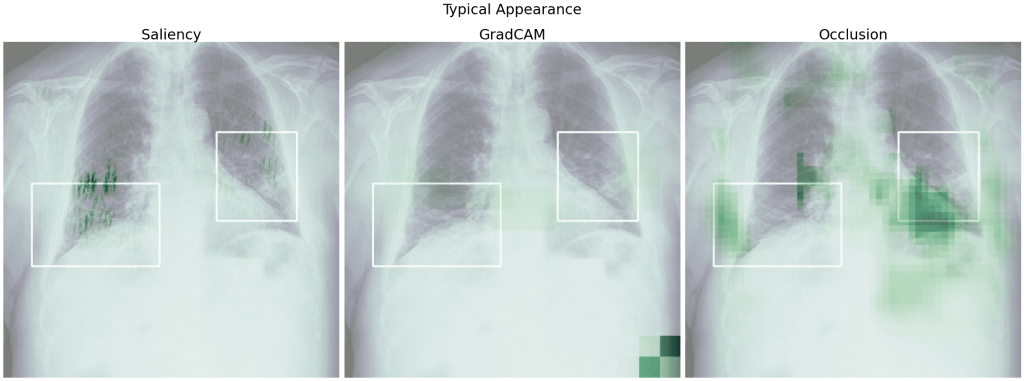
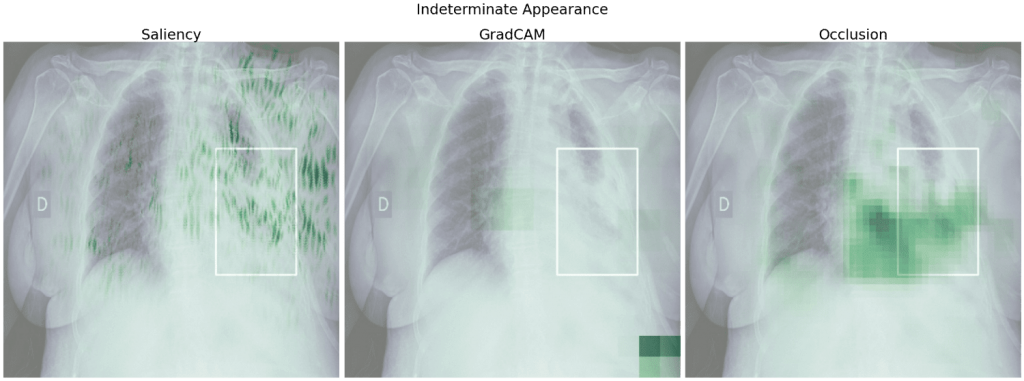
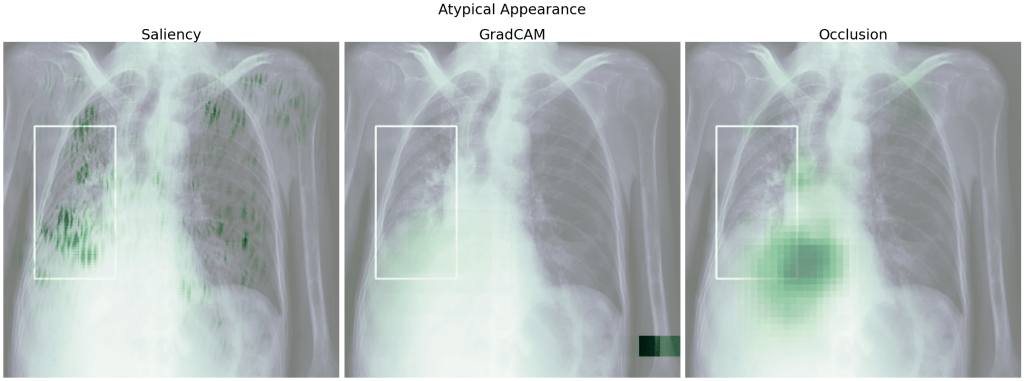
Widzimy, że w wielu przypadkach istotne dla predykcji były fragmenty płuc – tak jak tego oczekiwaliśmy i nawet pokrywają się częściowo z ramkami zaznaczonymi przez lekarzy. Mało informacji zdaje się przekazywać tutaj GradCAM – na każdym zdjęciu mamy zaznaczony prawy dolny róg i słabo zaznaczone pozostałe obszary.
Zobaczmy teraz heatmapy dla obrazu, w którym model źle określił klasę, przykładowo dla klasy Indeterminate Appearance przewidział Negative for Pneumonia. Poza heatmapami dla klasy prawdziwej, sprawdzimy także, które obszary obrazu wpłynęły na predykcję klasy Negative for Pneumonia.
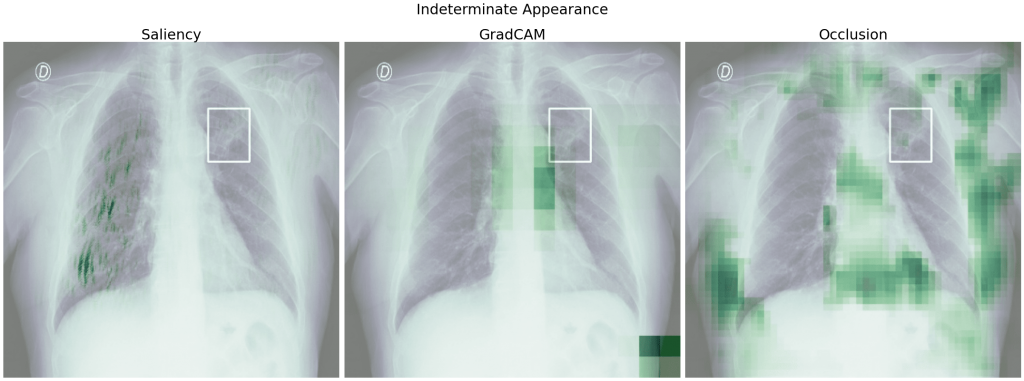
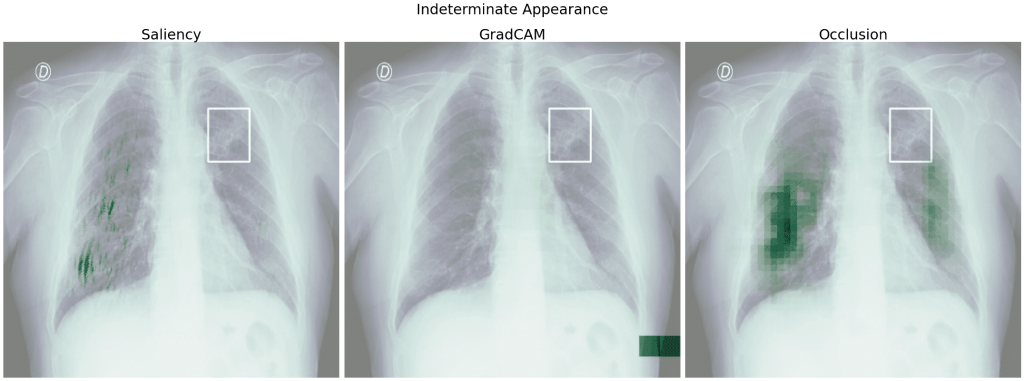
Dla klasy wygrywającej – Negative for Pneumonia – podobnie jak powyżej istotnymi obszarami obrazu są fragmenty płuc, z kolei dla klasy prawdziwej są to również obszary poza płucami, co jest szczególnie uwidocznione przy Occlusion. Gdybyśmy nie znali prawdziwej klasy, ufając bezkrytycznie modelowi i heatmapom, moglibyśmy niefortunnie uznać klasę Negative for Pneumonia za prawdziwą.
Spójrzmy teraz na heatmapy uzyskane względem klasy prawdziwej dla klasyfikatora binarnego (model 8). Dla porównania wybrałam te same zdjęcia jak dla modelu 5 (tak się złożyło, że model 8 dla tych zdjęć dał te same wyniki, tj. dla klasy Negative for Pneumonia przewidział Negative, a dla pozostałych klas Positive).
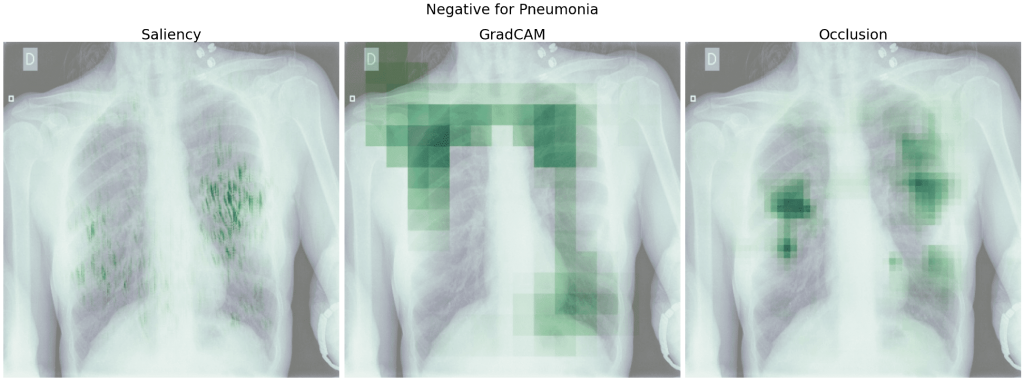
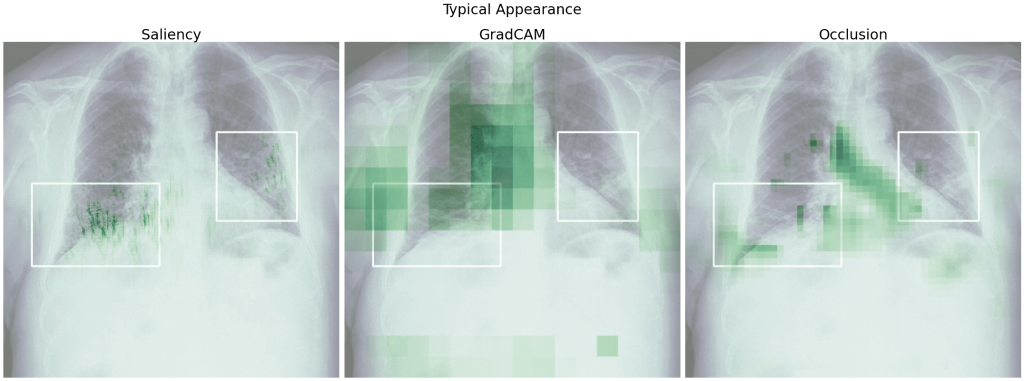
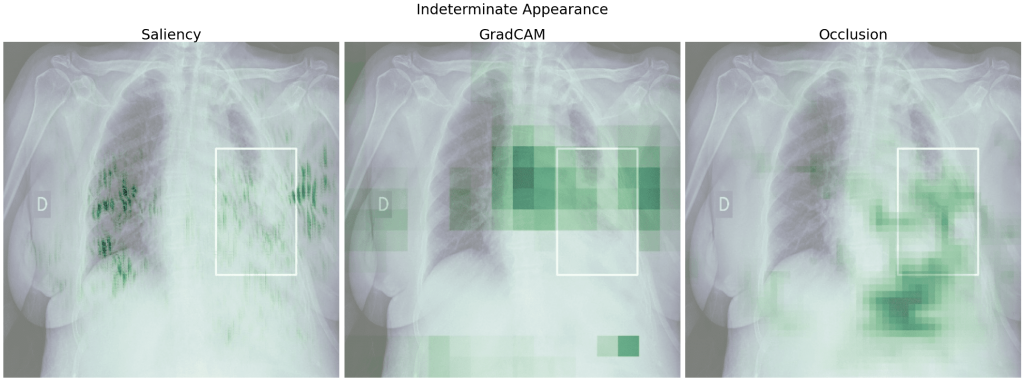
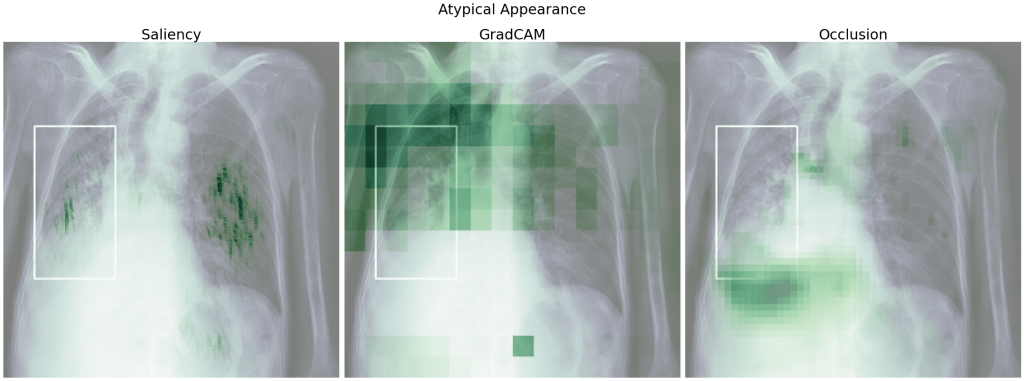
Podobnie jak dla modelu 5 w wielu przypadkach zaznaczone są płuca i istotne dla modelu obszary częściowo pokrywają się z ramkami. Więcej obszaru zaznacza tutaj GradCAM i nie mamy już 'szczególnie ważnego’ rogu obrazu.
Zobaczmy jeszcze heatmapy modelu 8 dla źle przypisanych klas (dla porównania również te same zdjęcia co powyżej):
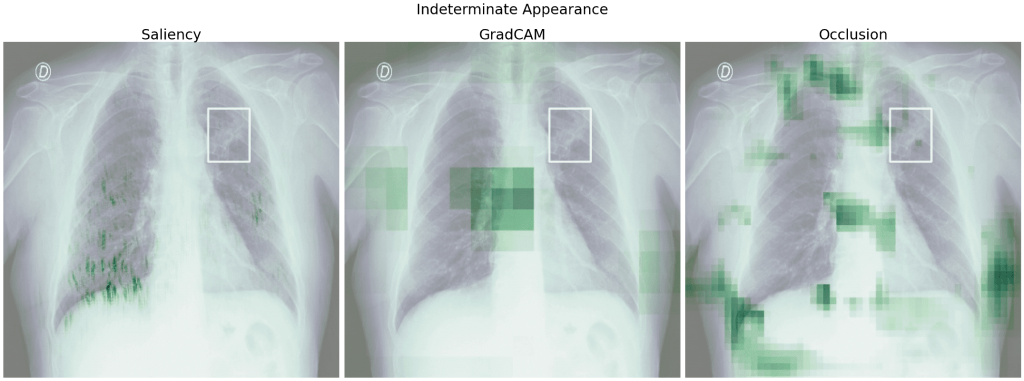
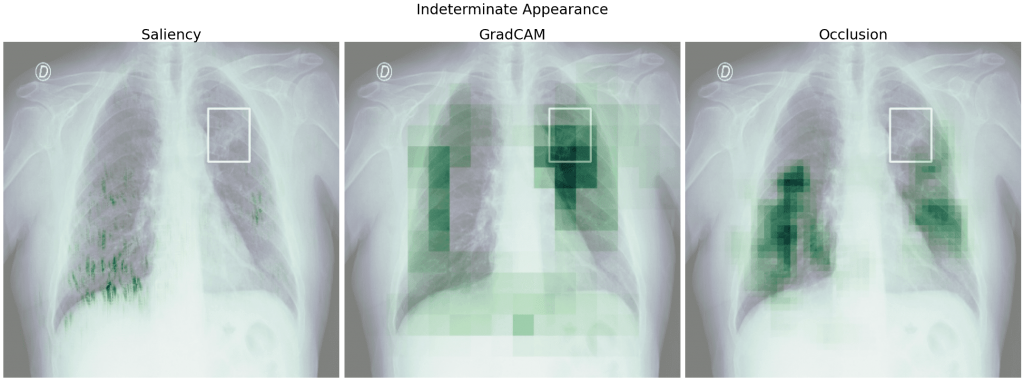
Tak jak dla modelu 5, dla modelu 8 dla klasy prawdziwej ważniejsze są obszary poza płucami, natomiast dla źle przewidzianej klasy obszary płuc.
Detekcja
Do detekcji użyłam YOLOv4 Darknet.
Dość dużym problemem okazało się uruchomienie Darknetu w środowisku Kaggle. Rozwiązaniem było użycie modułu Deep Neural Network biblioteki OpenCV, który umożliwia inferencję modeli z różnych frameworków, w tym właśnie Darknetu. W takim podejściu kryją się jednak pewne pułapki:
- Trening z channels=1 – dla jednej ze starszych wersji Darknetu jeden kanał był w porządku, jednak dawał złe wyniki podczas inferencji w OpenCV, z kolei po aktualizacji Darknetu dla obrazów w skali szarości wartości mAP podczas treningu były niepoprawne.
- Jeśli chcemy zmienić próg wykrywania obiektów (threshold, domyślnie 0.2) lub próg usuwania pokrywających się bounding boxów (NMS threshold, domyślnie 0.4), należy pamiętać, aby przed inferencją dodać do warstw [yolo] w pliku .cfg odpowiednie nowe wartości, np.:
thresh = 0.1
nms_thresh = 0.3
Oprócz dobierania thresholdów sprawdziłam jeszcze jak na wynik wpływa data augmentation, wyłączenie mozaiki i policzenie custom anchors. Poniższa tabelka zawiera część eksperymentów dla dwóch podejść – detekcji jednej klasy (opacity) i detekcji trzech klas (do połączenia z klasyfikacją binarną). Wszystkie modele mają rozmiar wejściowy 928×928 piskeli.
| liczba klas | channels=1 | dataaug | mosaic | custom anchors | mAP [%] | |
|---|---|---|---|---|---|---|
| 1 | 1 | ✓ | ✓ | 48.56 | ||
| 2 | 1 | ✓ | 24.95 | |||
| 3 | 1 | ✓ | ✓ | 46.53 | ||
| 4 | 1 | ✓ | ✓ | ✓ | ✓ | 41.68 |
| 5 | 3 | ✓ | 18.86 | |||
| 6 | 3 | 14.42 | ||||
| 7 | 3 | ✓ | ✓ | 16.95 | ||
| 8 | 3 | ✓ | ✓ | 17.84 | ||
| 9 | 3 | ✓ | ✓ | ✓ | 14.73 |
Najlepsze modele wybrane do obliczenia ostatecznego wyniku to model 3 i model 5. Trening modelu 3 został przerwany, ponieważ mAP malało, mimo że loss również malał i najlepsze wagi model miał i tak na początku treningu.
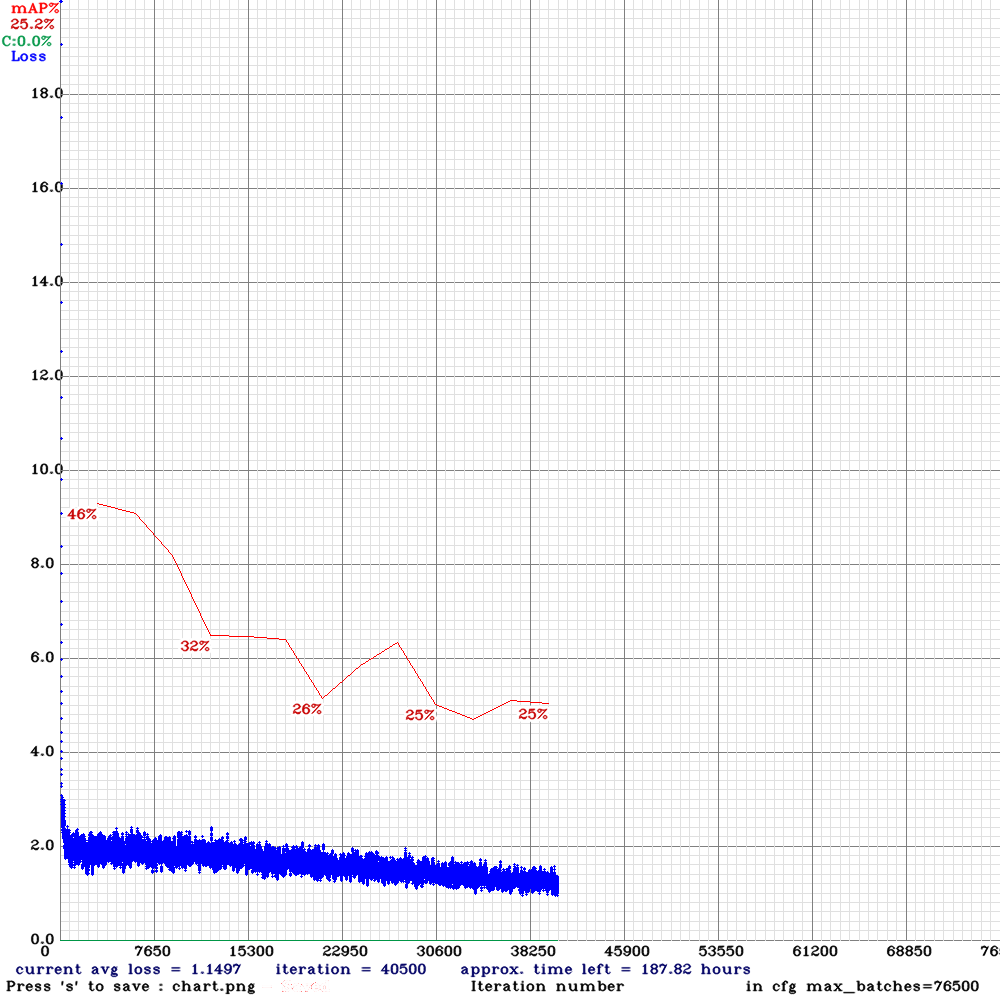
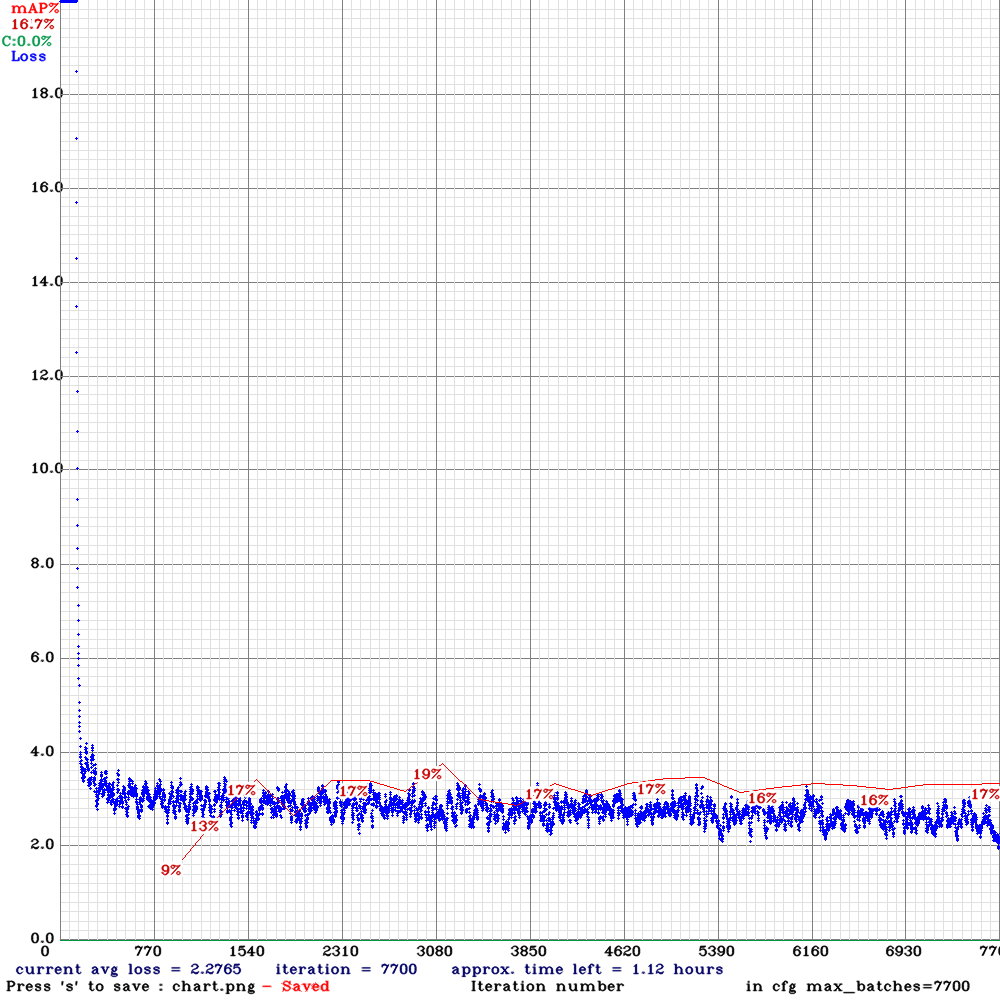
Spójrzmy na przykładowe predykcje obu modeli i ich krzywe Precision-Recall. Kolory na obrazach odpowiadają legendzie na wykresach. Na zielono są zaznaczone obiekty prawdziwe.
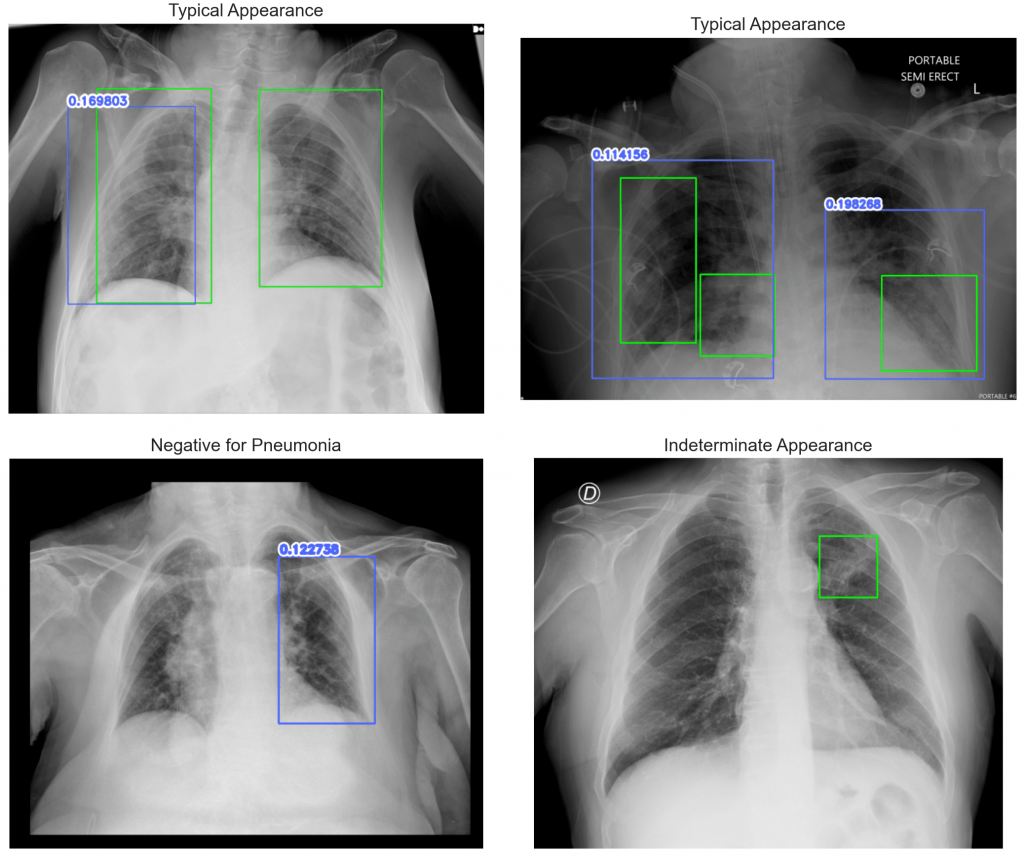
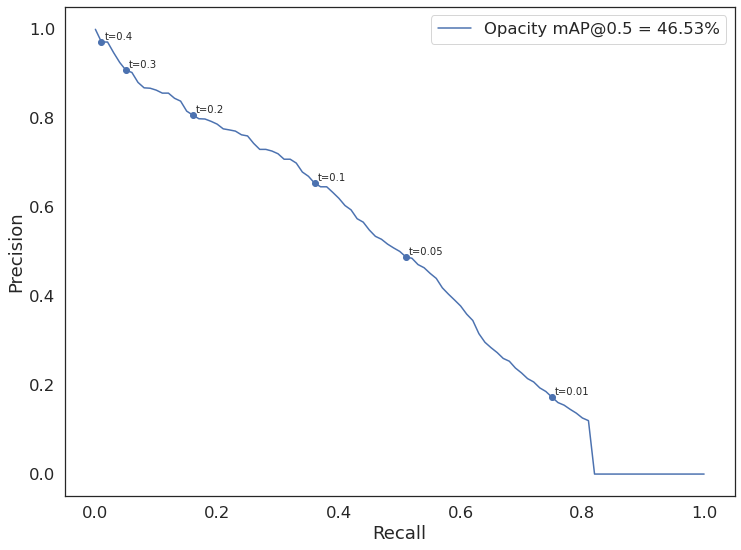
Widać na powyższych przykładach, że model radzi sobie średnio. Dla progu 0.1 mamy całkiem wysokie precision (> 0.6 – więcej niż połowa wykrytych obiektów jest poprawna), natomiast recall jest dość niski (< 0.4 – nie wykrywamy ponad połowy wszystkich prawdziwych obiektów; przypadki takie jak Indeterminate Appearance powyżej). Zdarzają się obiekty wykryte na obrazach Negative for Pneumonia, czasem model wykrywa jeden obiekt zamiast dwóch osobnych. Pocieszające jest, że wykryte obiekty w każdym przypadku znajdują się na obszarze płuc.
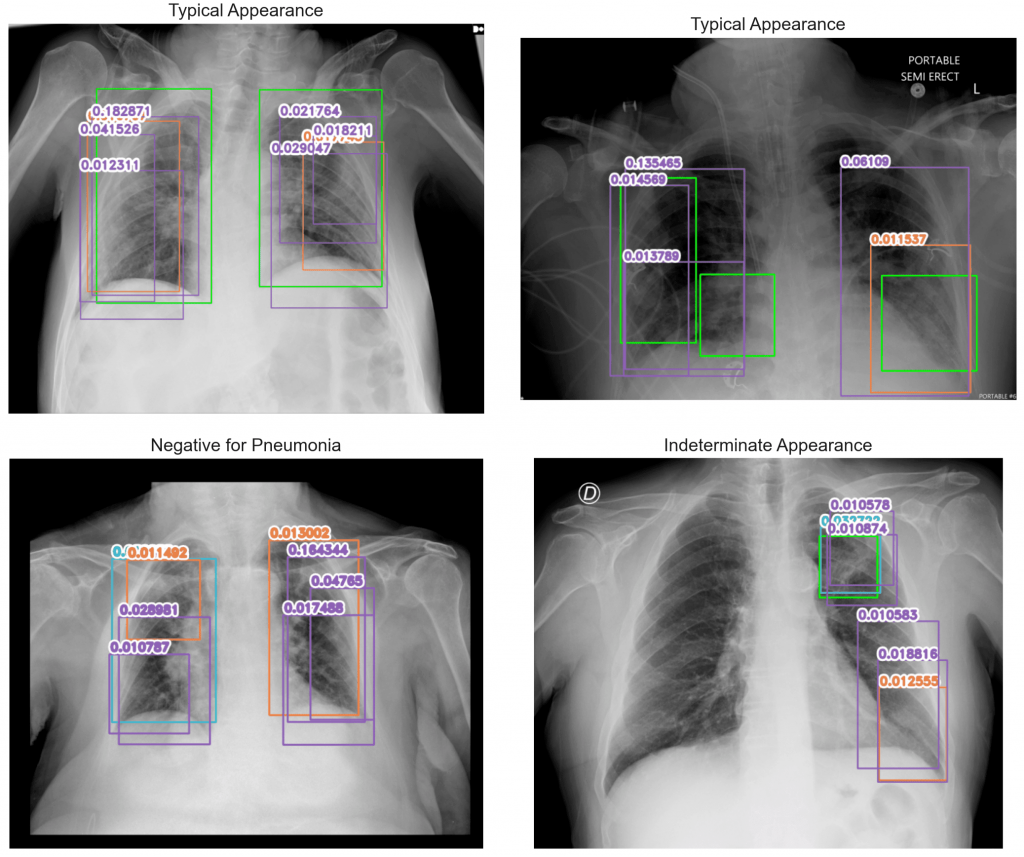
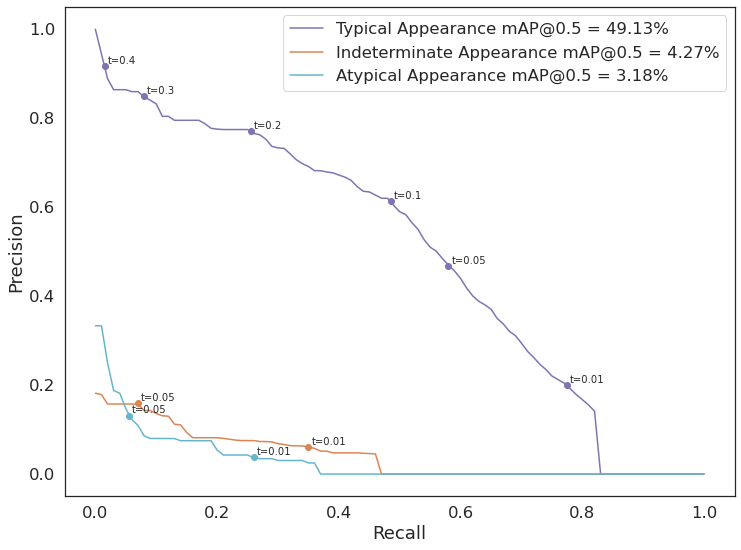
Bardzo niski threshold wybrany dla modelu 5 powoduje, że mamy dużo obiektów na każdym z obrazów. Widzimy też, że na danym obrazie występują obiekty różnych klas. Określając na tej podstawie klasę obrazu, wybierałam obiekt o najwyższym confidence. Z wykresu można odczytać, że dla progu 0.01 mamy wysoki recall (niemal 0.8, a więc mało obiektów prawdziwych nam umyka – niskie False Negatives), jednak precision jest niskie (około 0.2 – jest dużo obiektów wykrytych nadmiarowo – wysokie False Positives).
Podusmowanie
Dwa podejścia wybrane do policzenia ostatecznego wyniku to:
| Klasyfikator | Detektor | Private Score | Public Score |
|---|---|---|---|
| 5 | 3 | 0.404 | 0.410 |
| 8 | 5 | 0.370 | 0.379 |
Private Score jest liczony na podstawie ~71% danych testowych, a Public Score na podstawie ~29%. Ostateczny wynik nie zachwyca i daje miejsce 1026 (Private) oraz 1055 (Public). Dałoby się go jeszcze na pewno poprawić, zważywszy na to, że pierwsze miejsce w konkursie to był wynik: Private – 0.635, Public – 0.658.
Jednym z głównych mankamentów powstałych modeli – zarówno klasyfikatorów jak i detektorów – jest słaba rozróżnialność klas pozytywnych. Wynika to z mniejszej liczby zdjęć w zbiorze klas Indeterminate Appearance i Atypical Appearance, a być może i z ogólnego podobieństwa do siebie każdej z prezentacji COVID-19 i posiadania wspólnych cech na obrazie rentgenowskim.
Aby zmieścić się w ramach czasowych konkursu, część podejść należało niestety odrzucić na rzecz innych. Kilka pomysłów na poprawę wyników to:
- przycięcie obrazu do płuc – wytrenować model, który przycina obraz do płuc, dzięki czemu na obrazie pozostaje mniej nieistotnych z medycznego punktu widzenia informacji,
- wybranie innej architektury do klasyfikacji i/lub detekcji,
- ensemble – ostateczna predykcja jest wynikiem wielu modeli; wyniki poszczególnych modeli mogą być równoważne lub można nadać im wagi,
- cross validation – oceniać modele nie na podstawie wyników ze sztywno podzielonego zbioru na zbiór treningowy i walidacyjny, a na podstawie cross walidacji, tj. podzielić zbiór na zbiór treningowy i walidacyjny n razy, następnie wytrenować i przetestować dany model dla każdego podziału i obliczyć średnią uzyskanych wyników,
- wybrać/dodać inne techniki data augmentation, np. RandomErasing, RandomSunFlare.