W połowie maja zakończyła się druga edycja konkursu Shifts Challenge – Shifts Challenge 2022 zorganizowanego przez Shifts Project. Głównym celem challenge’u jest podnoszenie świadomości na temat problemu rozbieżności między danymi źródłowymi a docelowymi (ang. domain/distributional shift). Poza tym zwraca on także uwagę na wartość szacowania niepewności predykcji i zdolność generalizacji modeli podczas stosowania ich do danych rzeczywistych, w szczególności w zadaniach wysokiego ryzyka.
Jednym z tematów zadanych w ramach konkursu była segmentacja zmian patologicznych istoty białej na obrazach 3D rezonansu magnetycznego w przebiegu stwardnienia rozsianego. Razem z Karolem przystąpiliśmy do rozwiązania tego zadania i zajęliśmy 3 miejsce w ostatecznym rankingu. Nasz kod jest publicznie dostępny: https://github.com/deepdrivepl/shifts.
Wprowadzenie
Stwardnienie rozsiane (ang. Multiple Sclerosis, MS) jest przewlekłą chorobą ośrodkowego układu nerwowego, na którą na świecie choruje 2,9 miliona osób, a liczba ta od lat rośnie.
Choroba charakteryzuje się wieloogniskowym uszkodzeniem tkanki nerwowej. Obszary uszkodzenia istoty białej (ang. white matter lesions, WML) można uwidocznić w badaniu rezonansem magnetycznym – są one dobrze widoczne na obrazach FLAIR. Segmentacja WML pozwala na określenie ich liczby i rozmiaru, co jest istotne dla diagnozy choroby, oceny rokowania i monitorowania leczenia.
Do usprawnienia procesu segmentacji WML można wykorzystać modele uczenia maszynowego. Niestety żaden zbiór danych nie będzie na tyle reprezentatywny, aby uwzględniać wszystkie możliwe właściwości obrazów WML oraz warunki, w jakich powstały. Ta różnorodność obrazów – czyli wspomniany już domain shift – powoduje spadek jakości modeli i ogranicza ich zastosowanie w rzeczywistości. Jest ona spowodowana tym, że obrazy mogą pochodzić z różnych ośrodków, urządzeń, co za tym idzie są wykonywane przez różne osoby i przetwarzane przez różne oprogramowanie. Na shift wpływa również faza choroby (wielkość i liczba zmian) oraz demografia pacjentów (wiek, płeć). Uwzględnienie tych aspektów i stworzenie modelu o dobrej generalizacji przyczyniłoby się do poprawy jakości i wydajności opieki medycznej pacjentów ze stwardnieniem rozsianym.
Dane
Dane udostępnione w ramach challenge’u to obrazy FLAIR (z pozostałych modalności nie skorzystaliśmy) z odpowiadającymi im maskami: ground truth WML i mózgu. Wszystkie obrazy są w formacie NIfTI. Zostały podzielone na zbiór treningowy, dwa walidacyjne dev_in i eval_in oraz testowy dev_out. Przyrostek in („in domain”) oznacza, że nie występuje domain shift pomiędzy danym zbiorem, a zbiorem treningowym. Natomiast out („out of domain”) wskazuje, że jest shift pomiędzy zbiorem out a zbiorem treningowym i najprawdopodobniej na tym zbiorze zaobserwujemy pogorszenie metryk modelu w porównaniu do pozostałych zbiorów.
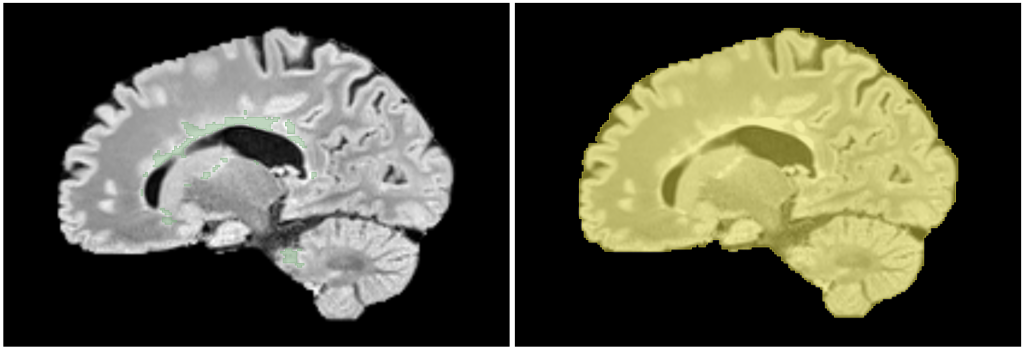
Udostępnione obrazy są już wstępnie przetworzone: zostały odszumione, usunięto czaszkę (co działa jako dodatkowa anonimizacja danych), przeprowadzono korekcję niejednorodności pola magnetycznego i interpolację do przestrzeni, w której woksel ma rozmiar 1 na 1 na 1 mm.
Zastrzeżenie: ze względów licencyjnych obrazy pojawiające się we wpisie pochodzą tylko ze zbioru dev_out.



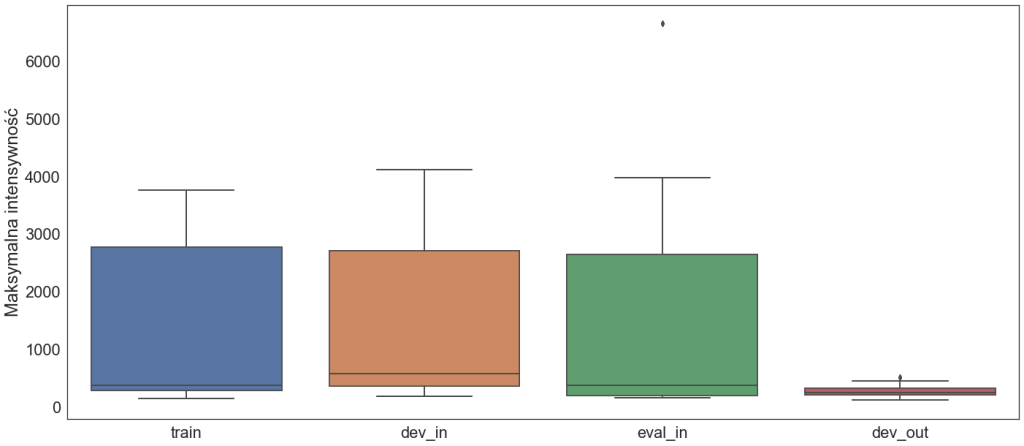
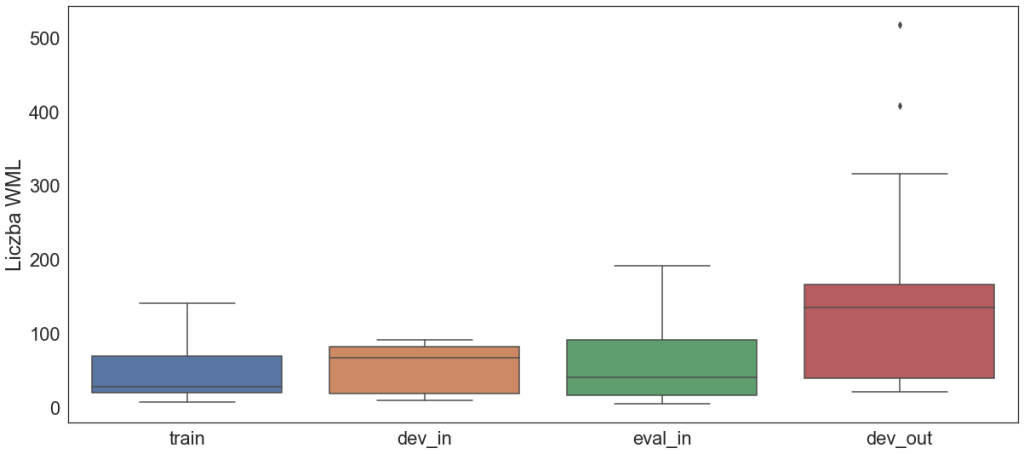
Przykłady shiftu pomiędzy podzbiorami można zobaczyć na poniższych wykresach. Zbiór dev_out różni się od pozostałych między innymi pod kątem maksymalnej wartości intensywności oraz liczby WML na jednym obrazie.
Eksperymenty
Punktem wyjścia był baseline udostępniony przez organizatorów. Przeszliśmy z PyTorcha na PyTorch Lightning i zaczęliśmy od treningów UNetu 3D. Po dopracowaniu pipeline’u zamieniliśmy model na bardziej wymagający: U²Net z transformerem z repozytorium XUnet. Ze względu na ograniczone zasoby transformer u nas znajdował się tylko w segmencie na końcu enkodera i segmencie na początku dekodera. Implementacja XUnetu jest elastyczna i można inicjować zwykły Unet lub U²Net, które też testowaliśmy, jednak bez nadzwyczajnych efektów.
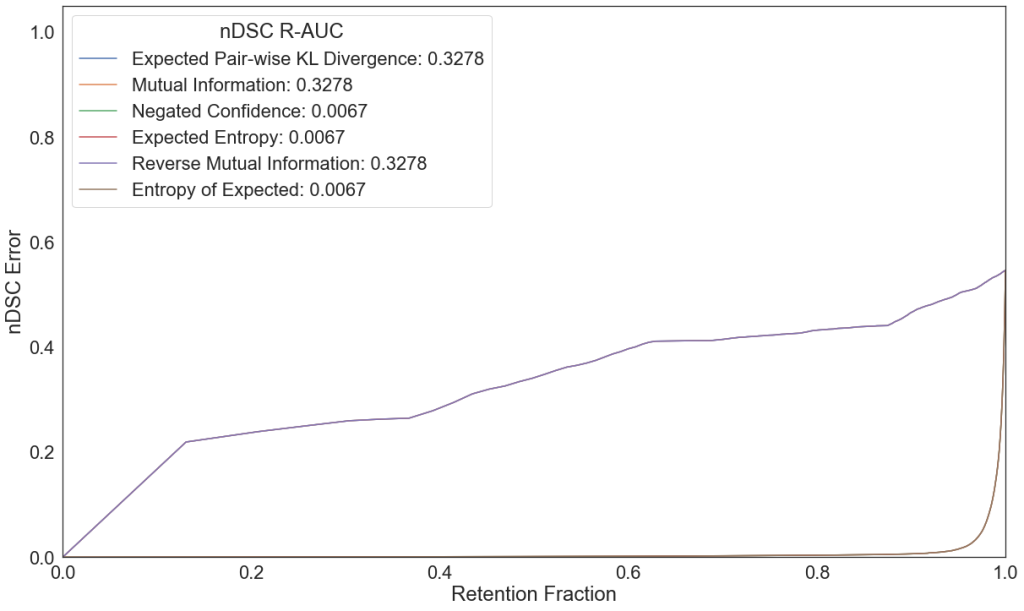
Metryką segmentacji w challenge’u był znormalizowany Dice. Natomiast metryką determinującą pozycję w rankingu było pole pod krzywą error retention (opis krzywych można znaleźć w artykule organizatorów). Do szacowania niepewności i konstruowania krzywej wykorzystaliśmy miary zaproponowane i zaimplementowane przez organizatorów.
Nasze eksperymenty i pomysły obejmowały m.in.:
- dobór learning rate i parametrów schedulera,
- skalowanie lossu przez współczynnik zależny od procentowego udziału WML w danym batchu,
- sprawdzanie, jakie transformacje data augmentation poprawiają wyniki, a także, czy stos transformacji poprawia u nas generalizację,
- klasyfikacja procentowego udziału WML w obrazie wejściowym na podstawie cech wydobytych przez enkoder i uwzględnienie tego elementu jako MSE w lossie całkowitym.
Funkcje kosztu
Jedną z serii eksperymentów była analiza różnych funkcji kosztu i ich kombinacji. Wybraliśmy kilka lossów typowo do segmentacji i kilka lossów dla klasyfikacji.
| Segmentacja | Klasyfikacja | Kombinacje |
| Dice | Focal | Dice + Focal |
| nDSC | CE | Generalized Dice + Focal |
| Log-Cosh Dice | BCE | nDSC + Focal |
| Tversky | nDSC + Focal + CE |
Zauważyliśmy, że wszystkie modele dają porównywalne wyniki segmentacji mierzone poprzez nDSC ze spodziewanym spadkiem metryki na zbiorze dev_out w porównaniu do zbiorów dev_in i eval_in.
Modele trenowane z lossem do segmentacji dają gorszą niepewność i tym samym większe pole pod krzywą error retention. Nie dotyczy to lossu nDSC, który dał porównywalne wyniki do lossów klasyfikacyjnych. Jest to związane z tym, że nasz loss nDSC pod spodem korzysta z binary cross entropy do policzenia składowych: loss true positives, loss false positives i loss false negatives. Dla wszystkich funkcji kosztu obserwujemy wzrost pola pod krzywą error retention dla zbioru dev_out.
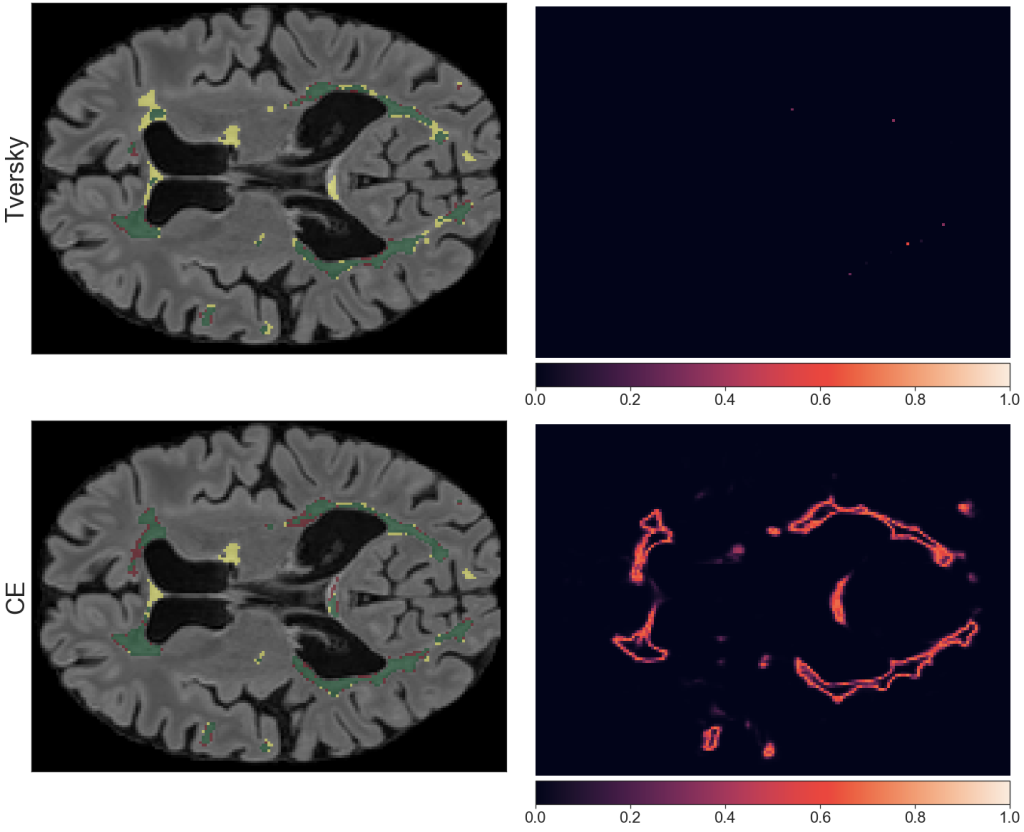
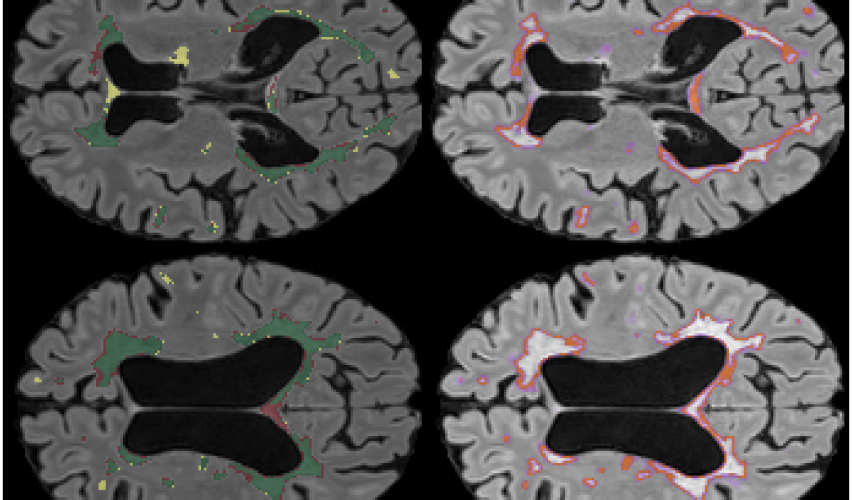
zielony – true positive, żółty – false negative, czerowny – false positive.
Rozwiązania finalne
Ostatecznie udało nam się zająć 3 i 4 miejsce. Ranking jest publiczny i można go zobaczyć pod tym adresem.
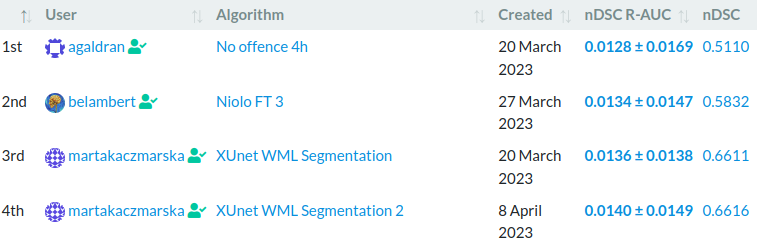
3 miejsce
Nasze 3 miejsce to model XUnet wytrenowany z nDSC jako funkcja kosztu. Pozostałe parametry treningu można zobaczyć w pliku konfiguracyjnym w naszym repozytorium. Inferencja jest wykonywana metodą sliding window z wielkością okna 64x64x64 i zakładką 0.25. Przed podaniem do modelu obraz jest normalizowany.
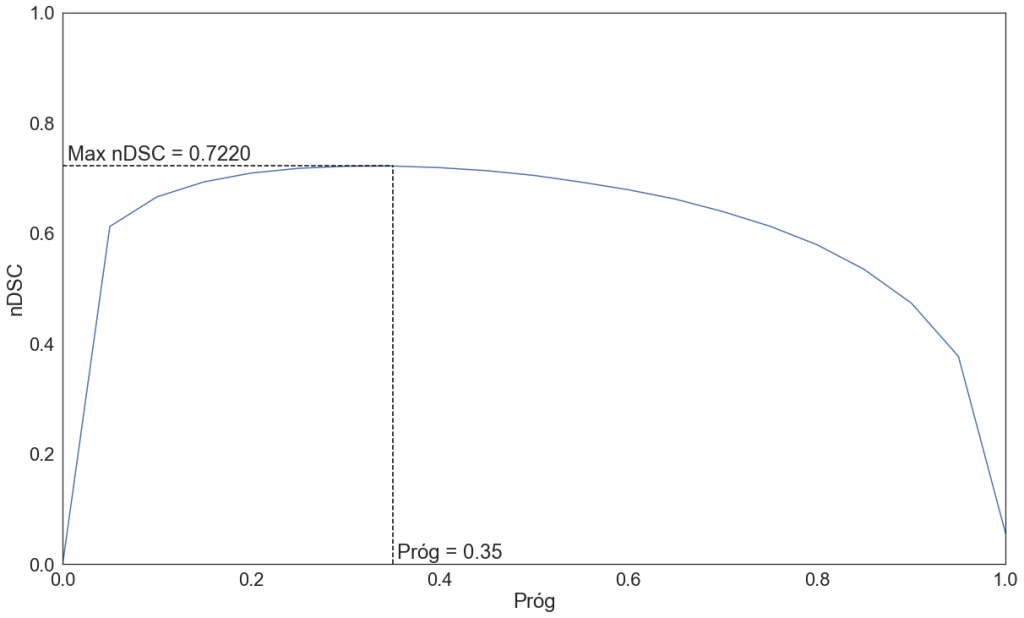
W celu uzyskania maski binarnej po funkcji sigmoid wynik jest progowany z progiem 0.35. Jest to wartość, dla której uzyskujemy maksymalny nDSC na zbiorze eval_in.
Jako miarę niepewności wybraliśmy entropy of expected, ponieważ daje małe pole pod krzywą error retention oraz opisuje niepewność całkowitą.
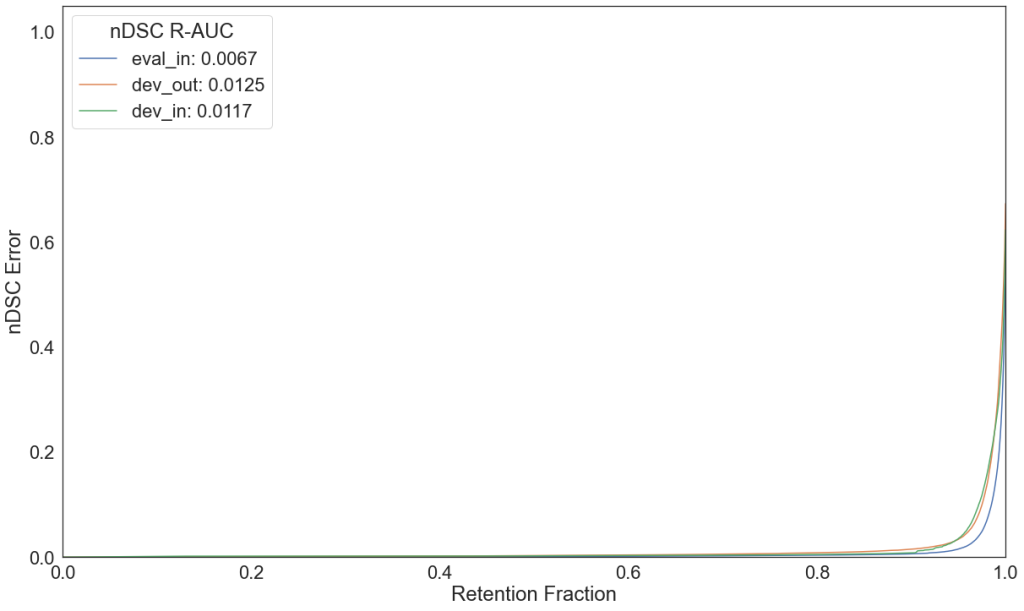
Małe pole pod krzywą sugeruje, że dana niepewność dobrze koreluje z błędami modelu. Uzyskana mapa niepewności pokazuje wątpliwe miejsca i daje informacje o niepewnych konturach WML oraz o false positives i false negatives.
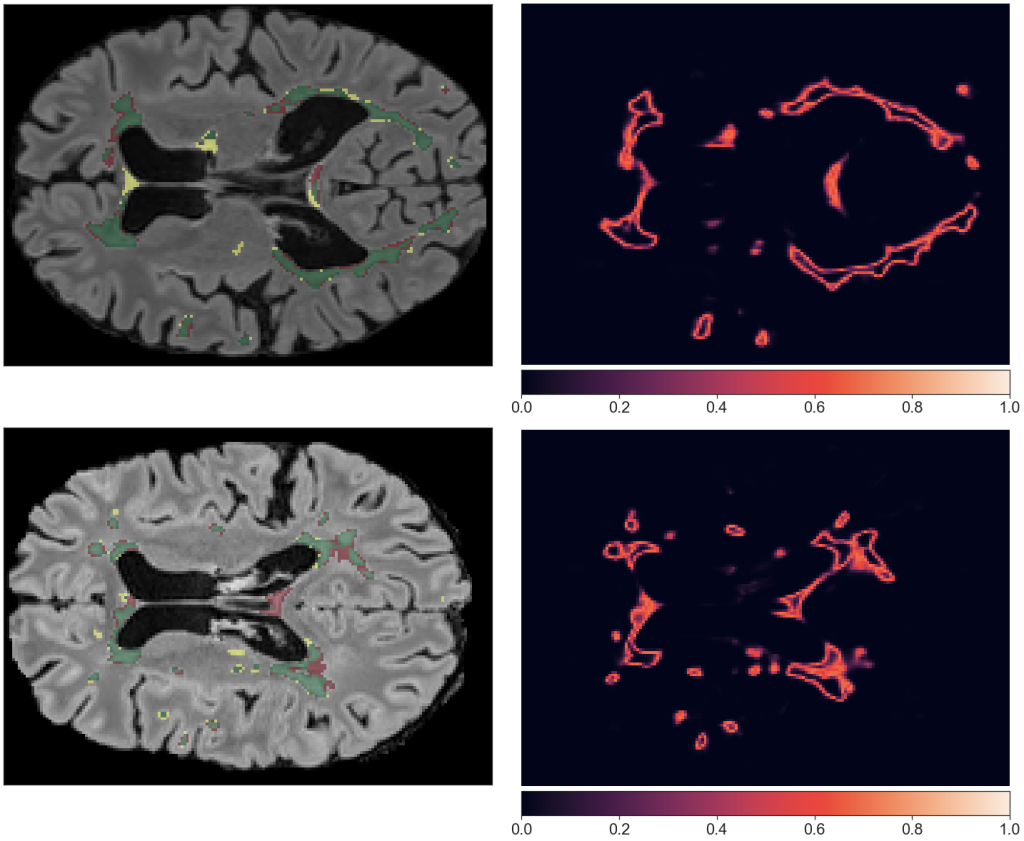
zielony – true positive, żółty – false negative, czerowny – false positive.
4 miejsce
Rozwiązanie z 4 miejsca było próbą asekuracji, gdyby submission z 3 miejsca nie spełniło wymagań odnośnie czasu inferencji. Ostatecznie niewykorzystaną, ponieważ nasze 3 miejsce zostało zaakceptowane.
Rozwiązanie 4. różni się od rozwiązania 3. processingiem podczas inferencji, mającym na celu przyspieszenie całości. Przed predykcją przycinane jest tło przy użyciu transformacji CropForeground.
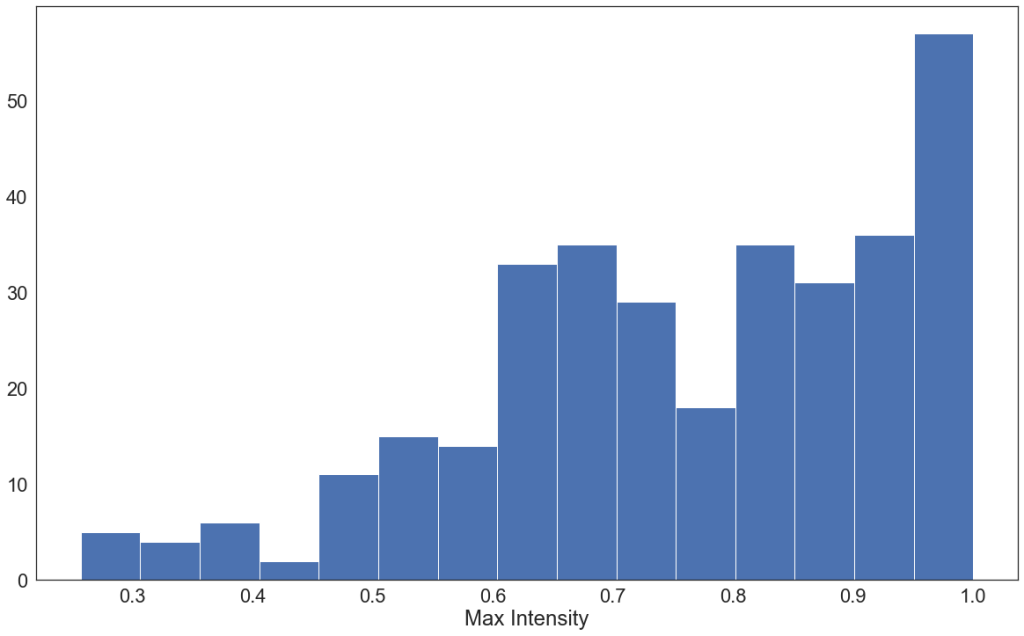
Następnie po normalizacji podczas wybierania patchy w metodzie sliding window sprawdzana jest maksymalna wartość intensywności danego patcha. Jeśli jest >0.2, to model normalnie wykonuje predykcję, a jeśli <=0.2, to taki patch jest pomijany i w predykcji zwracane są zera. Na koniec wykonywana jest transformacja odwrotna do CropForeground, aby uzyskać wynik o takim samym rozmiarze jak obraz wejściowy.
Wartość 0.2 wybraliśmy na podstawie analizy zbioru. Sprawdziliśmy maksymalne wartości intensywności patchy zawierających WML po normalizacji w każdym z podzbiorów. Histogramy zaczynały się około 0.3-0.4. Wybraliśmy wartość trochę niższą, aby wziąć pod uwagę domain shift.
Podsumowanie
Podczas challenge’u doświadczyliśmy trudności, jakie można napotkać przy tworzeniu rozwiązań do realnych zastosowań w medycynie:
- mały zbiór obrazów medycznych,
- wpływ domain shiftu na wyniki,
- wyjaśnialność predykcji.
Ostatnia kwestia – wyjaśnialność poprzez szacowanie niepewności – pozwala na zasygnalizowanie, kiedy model myli się lub kiedy może się mylić, co jest znaczące w praktyce. Lekarz otrzymuje dodatkowy output w postaci mapy niepewności, która pomaga w analizie i poprawie predykcji modelu. Przekłada się to na zwiększenie bezpieczeństwa systemu, ponieważ bezkrytyczne przyjmowanie podpowiedzi z automatu, gdy otrzymujemy teoretycznie jednoznaczny wynik, może prowadzić do spadku dokładności lekarzy i złej diagnozy.
Organizatorzy dostarczyli dobrze przygotowany zbiór danych, który może służyć innym jako benchmark dla segmentacji, gdzie domain shift jest znaczny, a zniwelowanie jego wpływu jest kluczowe.
Warto wypatrywać kolejnej edycji konkursu, ponieważ zwraca uwagę na istotne problemy pojawiające się przy tworzeniu rozwiązań mających skutecznie działać w rzeczywistości, a także dostarcza wartościowych danych do ewaluacji modeli, które muszą być odporne na domain shift oraz oceny jakości wybranych metod niepewności.
Linki
- https://github.com/deepdrivepl/shifts – repozytorium z naszym rozwiązaniem
- https://shifts.grand-challenge.org/ – strona challenge’u na platformie Grand Challenge
- Shifts 2.0: Extending The Dataset of Real Distributional Shifts – artykuł organizatorów zawierający zarys problemu, szczegółowy opis danych i ich wyniki
- Novel structural-scale uncertainty measures and error retention curves: application to multiple sclerosis – artykuł organizatorów o szacowaniu niepewności segmentacji zmian patologicznych w stwardnieniu rozsianym
- https://github.com/lucidrains/x-unet – repozytorium modelu XUnet
Na koniec odsyłam jeszcze do prezentacji, w której opowiadam o challenge’u oraz ogólnie na temat segmentacji medycznych obrazów 3D: