Anatomiczne płaszczyzny ciała człowieka są niezwykle ważne w radiologii, będące podstawą do przeprowadzenia prawidłowego badania. Bez tej wiedzy trudno o uzyskanie wiarygodnego skanu. O ile zagadnienie płaszczyzn anatomicznych jest modułem obowiązkowym na kierunkach medycznych, tak informatykom sprawia to niemałą trudność. Zwłaszcza tym, którzy zajmują się gałęzią informatyki medycznej. Dlatego w zespołach badawczych powinien być przynajmniej jeden lekarz radiolog, który pomoże rozwikłać tajemnice wnętrza ludzkiego ciała. W dzisiejszym wpisie o tym, jak ułatwić sobie wyznaczenie takich płaszczyzn i jakie korzyści niesie ze sobą używanie standardu DICOM.
Co to jest DICOM?
DICOM (Digital Imaging and Communications in Medicine) to standard stosowany w medycynie do przesyłania, przechowywania, drukowania i przetwarzania obrazów medycznych oraz danych związanych z ich wytwarzaniem i interpretacją. Służy do umożliwienia współpracy różnych urządzeń i oprogramowania medycznego, takich jak skanery rezonansu magnetycznego, aparaty rentgenowskie, aparaty USG itp. Dzięki DICOM obrazy medyczne mogą być przesyłane między różnymi urządzeniami i systemami informatycznymi, co umożliwia ich wymianę i udostępnianie lekarzom i innym pracownikom medycznym.
Format DICOM został opracowany przez National Electrical Manufacturers Association (NEMA) przy współpracy z American College of Radiology (ACR), z potrzeby stworzenia sposobu, który pozwoliłby na odczyt jednego badania na wielu urządzeniach. Przed jego stworzeniem, powszechnym problemem występującym w środowisku medycznym była utrudniona interpretacja wyników badań, która była zależna od interpretacji zaleceń sprzętowych producentów.
Korzyści ze stosowania formatu DICOM i czym różnią się od tradycyjnych obrazów
Standaryzacja sposobu zapisu danych umożliwiła odczyt przez praktycznie każdy sprzęt medyczny. Dlatego wyniki badania tomografii komputerowej czy rezonansu magnetycznego mogą być odczytywane i analizowane w ramach jednej stacji diagnostycznej. Standaryzacja komunikacji pomiędzy urządzeniami umożliwiła również możliwość tworzenia systemów transmisji oraz archiwizacji danych obrazowych. Mowa oczywiście o systemach PACS (Picture Archiving and Communication System).
Pliki JPG zawierają pojedynczy obraz, w kolorze lub w skali szarości. Ich rozmiar jest stosunkowo niewielki, co już powinno być olbrzymim plusem przy tak dużej liczbie danych wytwarzanych w ochronie zdrowia. Niestety ten format ma pewne ograniczenia, co nie można powiedzieć o standardzie DICOM. W ochronie zdrowia zaszła konieczność przechowywania dużego zbioru danych, np. na temat pacjentów. Pliki DICOM posiadają dużą objętość, dlatego wymagają specjalistycznego oprogramowania i odpowiedniego sprzętu komputerowego. W warunkach domowych do odtworzenia obrazu zapisanego w formacie .dcm wystarczy przeglądarka DICOM, np. MicroDicom lub RadiAnt (chociaż to oprogramowanie działa w wersji darmowej na określony czas).
Gdzie są wykorzystywane pliki DICOM?
Z DICOM oczywiście spotykamy się w sektorze ochrony zdrowia, a dokładnie w przypadku takich badań obrazowych jak:
- tomografia komputerowa (CT),
- rezonans magnetyczny (MRI),
- pozytonowa tomografia emisyjna (PET CT),
- badanie USG (ultrasound) i wiele innych.
Pliki DICOM znajdują zastosowanie, gdy wynik badania jest eksportowany na zewnętrzny nośnik, np. płytę CD/DVD. Obecnie każdy pacjent po wykonaniu badania otrzymuje na własny użytek płytę CD/DVD z jego wynikami zapisanymi w formacie DICOM. Na takiej płycie znaleźć można kilkanaście folderów zawierających wszystkie niezbędne dane. Dane te w takiej postaci mogą zostać również przekazane do analizy innemu specjaliście.
Wizualizacja i analiza obrazów DICOM w Pythonie
Na potrzeby tego wpisu stworzyłam kompletny przewodnik krok po kroku jak stosować metody i techniki przetwarzania obrazów do obrazów medycznych, aby w pełni zwizualizować dane jako obraz dwuwymiarowy. Zacznijmy jednak od podstaw. Potrzebna nam będzie biblioteka pydicom, którą można zainstalować z poziomu Anacondy.
Dla pip:
pip install pydicom
Dla Anacondy:
conda install -c conda-forge pydicom
Potrzebna będzie nam też baza obrazów, która będzie zawierała obrazy DICOM. Można skorzystać ze zbiorów znajdujących w bazie danych Fumpe, np. https://figshare.com/articles/dataset/Patient31/6265655?backTo=/collections/FUMPE/4107803.
Możemy podejrzeć, jak wygląda pierwsze zdjęcie z pobranego datasetu. Plik otwieramy za pomocą MicroDicom.
Tworzymy plik Pythona i sprawdzamy poprawność wcześniejszych działań. Podczas pisania tego artykułu używałam Visual Studio Code.
import pydicom as dicom
import matplotlib.pyplot as plt
path="./PAT031/D0001.dcm"
dcm = dicom.dcmread(path)
plt.imshow(dcm.pixel_array, cmap=plt.cm.gray)
plt.show()
Uzyskujemy taki oto obraz:
Możemy uzyskać metadane zapisane na obrazie:
Dodajemy w tym celu:
dcm = dicom.dcmread(path)
print(dcm)
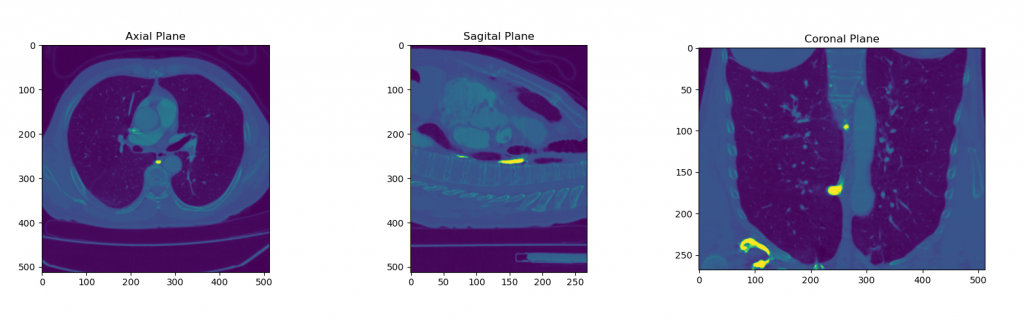
Wykreślanie płaszczyzny osiowej, czołowej i strzałkowej na obrazie DICOM w Pythonie
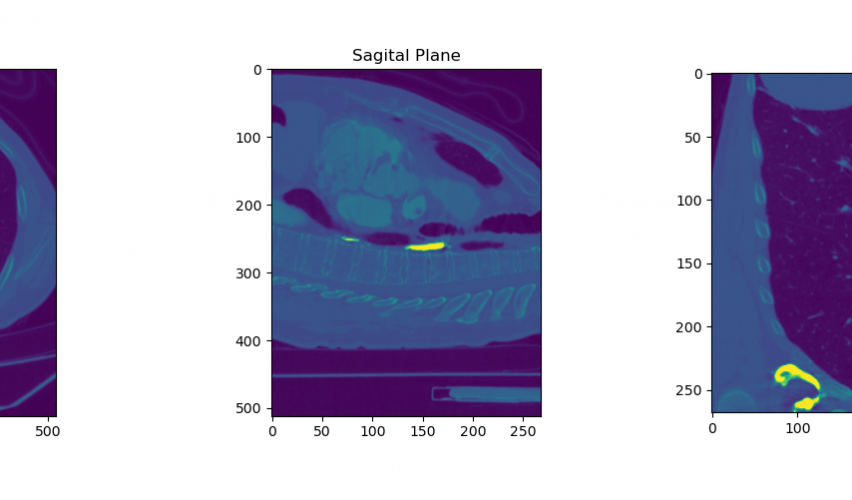
Zajmiemy się wykreśleniem płaszczyzny osiowej, koronalnej i strzałkowej na obrazie w Pythonie. Płaszczyzna strzałkowa jest płaszczyzną podłużną, dzielącą ciało na część prawą i lewą. Nie muszą one być równe, ale jeśli są równe, to płaszczyznę określa się jako midsagittal lub median plane. Płaszczyzna koronalna jest płaszczyzną podłużną, dzielącą ciało na część przednią (przód) i tylną (tył). Płaszczyzna osiowa (lub poprzeczna) to płaszczyzna pozioma dzieląca ciało na część górną (górną) i dolną (dolną).
Należy wybrać spójną płaszczyznę osiową do obrazowania mózgu, aby umożliwić powtarzalne pozyskiwanie i porównywanie obrazów. W przeciwieństwie do płaszczyzny strzałkowej, która jest nieodłącznie określona przez naszą wrodzoną lewoprawą płaszczyznę symetrii, płaszczyzny osiowa i koronowa wymagają uzgodnienia i na przestrzeni lat zaproponowano wiele definicji każdej z nich.
źródło: https://en.wikipedia.org/wiki/Anatomical_plane#/media/File:Human_anatomy_planes,_labeled.svg
Zacznijmy od wyświetlenia podstawowych informacji o pierwszym pliku.
import os
import pydicom as dicom
import numpy as np
import matplotlib.pyplot as plt
path = "./PAT031"
ct_img = os.listdir(path)
slices = [dicom.read_file(path + "/" + s,force=True) for s in ct_img ]
print(slices)
Powinniśmy otrzymać coś takiego:
Wykreślanie płaszczyzny osiowej
Zacznijmy od obliczenia pożądanych przez nas parametrów: axial, sagital i coronal (sagital i coronal przydadzą się później). Na współczynnik kształtu składają się zwykle dwie liczby całkowite. Wartości te nie reprezentują rzeczywistych szerokości i wysokości, a proporcję między wysokością a szerokością. Stosunek osiowy, dla dowolnej struktury lub kształtu o dwóch, lub więcej osiach, jest stosunkiem długości (lub wielkości) tych osi do siebie-dłuższa oś podzielona przez krótszą. W tym przypadku dzielimy pobraną wartość wartości pomiędzy pikselami (patrz: Rys. 4): [0.673828, 0.673828], otrzymując 1.
slices = sorted(slices, key = lambda x:x.ImagePositionPatient[2])
pix_spacing = slices[0].PixelSpacing
slices_thickness = slices[0].SliceThickness
axial_aspect_ratio = pix_spacing[1] / pix_spacing[0]
sagital_aspect_ratio = pix_spacing[1] / slices_thickness
coronal_aspect_ratio = slices_thickness / pix_spacing[0]
print("Pixel spacing:", pix_spacing)
print("Slice Thickness:", slices_thickness)
print("Axial Aspect Ratio:", axial_aspect_ratio)
print("Saital Aspect Ratio:", sagital_aspect_ratio)
print("Coronal Aspect Ratio:", coronal_aspect_ratio)
image_shape = list(slices[0].pixel_array.shape)
image_shape.append(len(slices))
volume_3d = np.zeros(image_shape)
#convert value into array
for i,s in enumerate(slices):
array_2D = s.pixel_array
volume_3d[:,:,i] = array_2D
print(array_2D.shape)
print(volume_3d.shape)
axial = plt.subplot(2,3,1)
plt.title("Axial Plane")
plt.imshow(volume_3d[:,:,image_shape[2] // 2])
axial.set_aspect(axial_aspect_ratio)
plt.show()
W ten sposób otrzymujemy obraz wynikowy.
Wykreślanie płaszczyzny strzałkowej
Płaszczyzna strzałkowa dzieli ciało lub organ na dwie części: prawą (dexter) i lewą (sinister). Wyznaczona jest przez oś strzałkową (axis sagittalis) i oś pionową (axis verticalis). Nazywa się ją również płaszczyzną pośrodkową lub płaszczyzną symetrii, ponieważ dzieli ciało na dwie symetryczne połowy: lewą i prawą. Równolegle do płaszczyzny strzałkowej można też poprowadzić dalsze płaszczyzny strzałkowe. W stosunku do płaszczyzny strzałkowej mówimy o kierunku przyśrodkowym (medialis) i bocznym (lateralis). Jeden z narządów może leżeć bardziej przyśrodkowo, a drugi bardziej bocznie. Może też mieć powierzchnię przyśrodkową i boczną. Jeżeli narząd położony będzie w płaszczyźnie strzałkowej, to będzie on zajmował położenie przyśrodkowe (medianus).
sagital = plt.subplot(2,3,2)
plt.title("Sagital Plane")
plt.imshow(volume_3d[:, image_shape[1] // 2, :])
sagital.set_aspect(sagital_aspect_ratio)
Pamiętaj, aby ta część kodu znalazła się przed:
plt.show()
Wykreślanie płaszczyzny czołowej (koronalnej)
Płaszczyzna czołowa (czasami nazywana też koronalną, od łacińskiego corona, „wianek, korona”) to dowolna pionowa płaszczyzna, która dzieli ciało na sekcje brzuszne i grzbietowe (brzuch i plecy). Płaszczyzna czołowa jest przykładem płaszczyzny podłużnej , ponieważ jest prostopadła do płaszczyzny poprzecznej. Dla człowieka płaszczyzna środkowa-koronowa przecinałaby stojące ciało na dwie połowy (przód i tył lub przednią i tylną) w wyimaginowanej linii, która przecina oba ramiona. Opis płaszczyzny czołowej dotyczy większości zwierząt, a także ludzi, mimo że ludzie chodzą wyprostowani, a różne płaszczyzny są zwykle pokazane w orientacji pionowej.
W tej sekcji dodajemy tylko instrukcje, które wyświetlą nam obraz w innej płaszczyźnie.
coronal = plt.subplot(2,3,3)
plt.title("Coronal Plane")
plt.imshow(volume_3d[image_shape[0] // 2, :,:].T)
coronal.set_aspect(coronal_aspect_ratio)
Uzyskane obrazy
Uzyskano obrazy w trzech przestrzeniach:
Technologia dała nam możliwość zajrzenia do wnętrza ludzkiego ciała. Dzięki Wilhelmowi Röntgenowi obrazowanie medyczne wskoczyło z biegiem lat na bardzo wysoki poziom. Dało to możliwość postawienia dokładniejszej diagnozy, a najnowsze osiągnięcia naukowców z zakresu informatyki medycznej daje możliwość zmniejszenia daki promienionia podawanej podczas badania CT. To niesie pewną nadzieję, że obrazowanie medyczne wzniesie na poziom, który do tej pory był poza zasięgiem naszego wzroku.
Kod źródłowy
https://github.com/deepdrivepl/axial-coronal-sagital-plane-ct
Bibliografia
- Bidgood WD Jr, Horii SC, Prior FW, Van Syckle DE. Understanding and using DICOM, the data interchange standard for biomedical imaging. J Am Med Inform Assoc. 1997 May-Jun;4(3):199-212. doi: 10.1136/jamia.1997.0040199. PMID: 9147339; PMCID: PMC61235
- Qiangqiang Gu, Naresh Prodduturi, Jun Jiang, Thomas J. Flotte, Steven N. Hart, Dicom_wsi: A Python Implementation for Converting Whole-Slide Images to Digital Imaging and Communications in Medicine Compliant Files, Journal of Pathology Informatics, Volume 12, Issue 1, 2021, 21, ISSN 2153-3539, https://doi.org/10.4103/jpi.jpi_88_20
- Amir Arabi, Toktam Shahraki, Rezvan Nezam-slami, Hamed Esfandiari, Mehdi Tavakoli, Homayoun Nikkhah, Axial or coronal CT scan; which is more accurate in detection of open globe injury?, Injury, Volume 52, Issue 9, 2021, p. 2611-2615, ISSN 0020-1383, https://doi.org/10.1016/j.injury.2021.04.034
- Bochenek A., Reicher M., Anatomia człowieka, tom I, Wydawnictwo Lekarskie PZWL, Warszawa 2004
- Bartłomiej Sokołowski, Teresa Gwardjak, Anatomia człowieka – kompendium, Kraków, 2012, AWF Kraków, wyd. 1
- Dokumentacja pydicom – https://pydicom.github.io/pydicom/stable/
- Masoudi, Mojtaba; Saadatmand-Tarzjan, Mahdi (2018): FUMPE. figshare. Collection. https://doi.org/10.6084/m9.figshare.c.4107803.v1