Standardową metodą wykrywania wirusa SARS-CoV-2 jest reakcja łańcuchowa polimerazy z odwrotną transkrypcją (RT-PCR). Jednak w zależności od fazy choroby może dawać wyniki fałszywie negatywne. Uzupełniającą metodą w diagnostyce koronawirusa jest tomografia komputerowa, która według niektórych badań ma wyższą czułość niż RT-PCR.
COVID-19 na obrazach tomografii komputerowej może ujawniać się w podobny sposób co zapalenie płuc wywołane innymi wirusami. Posiada jednak również cechy charakterystyczne, co pozwala na odróżnienie od niektórych innych chorób układu oddechowego, chociaż nie zawsze jest to łatwe, na przykład w przypadku grypy.
W tym wpisie zajmiemy się analizą obrazów tomografii komputerowej klatki piersiowej oraz ich klasyfikacją do jednej z trzech klas. Porównanych zostanie kilka architektur. Mimo że COVID-19 może powodować również inne zmiany widoczne na obrazach CT, np. zakrzepicę, skupimy się głównie na zmianach patologicznych w płucach, gdyż to one są podstawą do diagnozy w oparciu o obraz z tomografu komputerowego (wiele prac bazuje właśnie na wysegmentowanych płucach do klasyfikacji/segmentacji zmian patologicznych, o czym można przeczytać m.in. w tym artykule przeglądowym). Spróbujemy również odpowiedzieć na pytanie, czy transfer learning z użyciem modeli pretrenowanych na ImageNet ma sens w przypadku obrazów medycznych.
Projekt można znaleźć na GitHubie.
Analiza zbioru
Zbiór danych znajduje się na GitHubie i jest już podzielony na zbiór treningowy, walidacyjny i testowy. Do zbioru testowego na razie nie zaglądamy i go nie analizujemy, aby uniknąć obciążenia związanego z podglądaniem danych (data snooping bias) i nie próbować dostosowywać skuteczności modelu specjalnie pod ten zbiór (choćby nieświadomie), co utrudniłoby generalizację na obrazy spoza zbioru.
Niektóre obrazy są w formacie jpg, niektóre png. Obrazów w zbiorze treningowym jest 4404, natomiast walidacyjnym 1341. Większość obrazów ma rozdzielczość 512 x 512 px, jednak zdarzają się również obrazy większe.
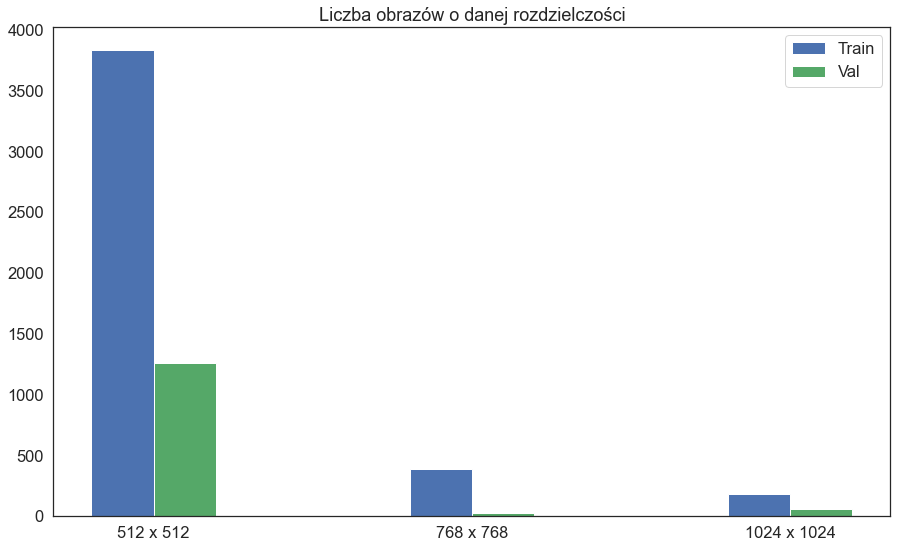
Są trzy klasy:
- COV – pacjent chory na COVID-19,
- Normal – zdrowy pacjent,
- OtherPneumonia – zapalenie płuc innego pochodzenia.
Zbiór nie jest zbalansowany – obrazów COV jest znacznie więcej niż pozostałych.
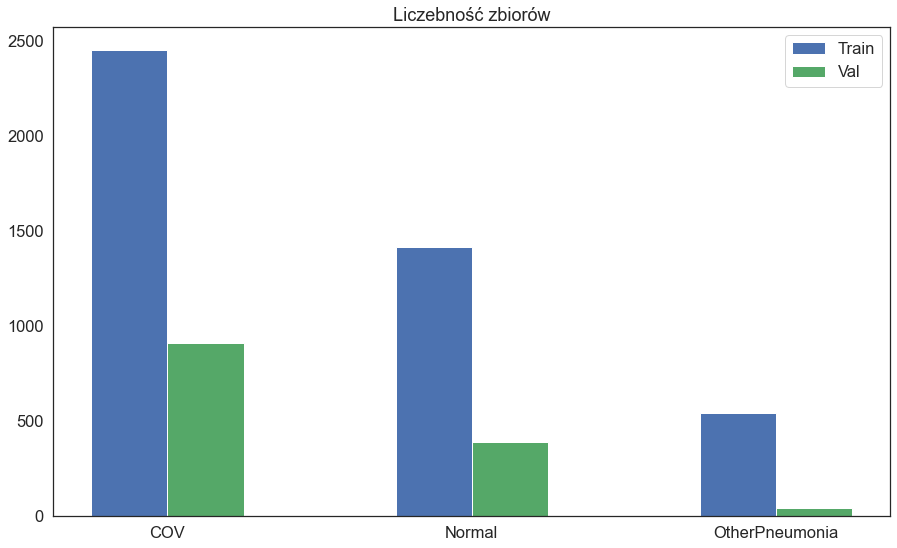
Niestety nie mamy więcej informacji na temat zbioru, na przykład czy obrazy pochodzą z jednego skanera, ilu pacjentów jest w zbiorze, jacy są to pacjenci, jakie dobrano okno do prezentacji skali szarości (ang. windowing). Brak tych oraz innych szczegółów na temat zbioru wpływa na jakość analizy, a także na interpretowalność uzyskanych modeli. Należy to mieć na uwadze przy projektowaniu i tworzeniu rozwiązań z użyciem deep learningu dla medycyny (pomocna w tym jest ta checklista).
Wizualizacje
Zwizualizujmy po kilka przykładów z każdej klasy:
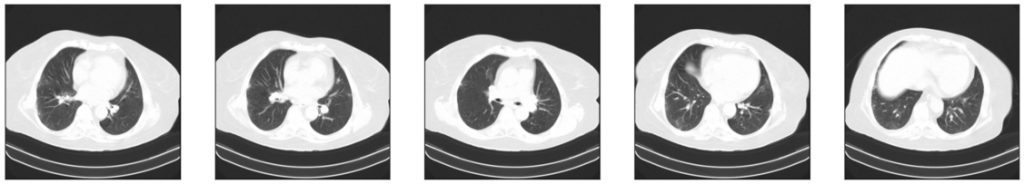
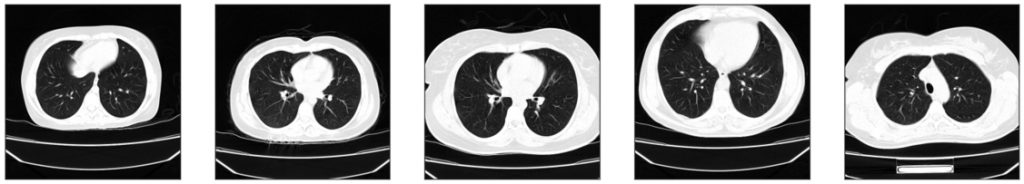
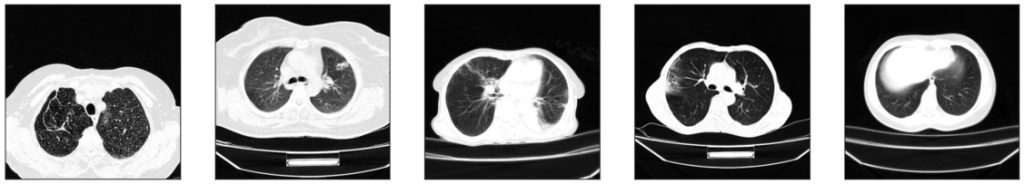
Na podstawie przykładowych obrazów widać, że w zbiorze może znajdować się więcej niż jeden przekrój od danego pacjenta. Widzimy również, że na zdjęciach oprócz pacjenta może znajdować się stół, a także że klatka piersiowa może zajmować mniej lub więcej powierzchni (tj. może być więcej lub mniej tła, które nie jest istotne przy klasyfikacji) i nie zawsze znajdować się dokładnie na środku obrazu.
Średni obraz
Obrazy w folderach Train oraz Val znajdują się w podkatalogach. Zobaczmy, czy poszczególne obrazy różnią się bardzo między sobą, wizualizując średni obraz z każdego podkatalogu. Poniżej znajdują się wizualizacje średnich obrazów zbioru treningowego i walidacyjnego. Poszczególne tytuły oprócz nazwy klasy zawierają nazwę katalogu oraz ile obrazów w danym katalogu się znajduje.
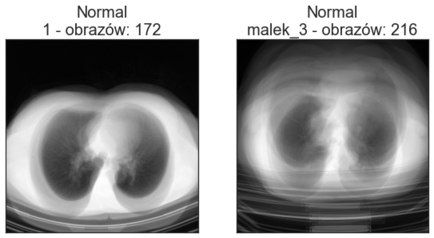
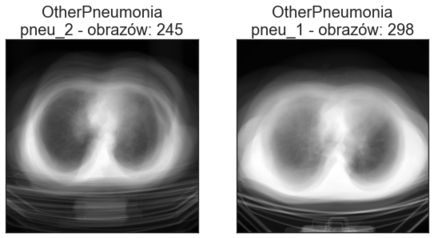
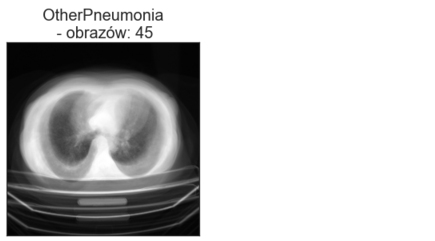
W obrębie danego podkatalogu obrazy nie zawsze są do siebie podobne pod względem wielkości klatki piersiowej oraz położenia na obrazie. Warto to mieć na uwadze podczas wstępnego przetwarzania obrazów. Przykładowo: jeśli chcielibyśmy usunąć z obrazu stół lub część tła, poszczególne obrazy wymagałyby różnych parametrów przycięcia (crop).
Zobaczmy również obraz średni ze wszystkich obrazów dla poszczególnych klas:
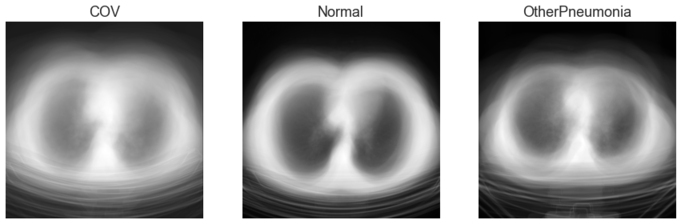
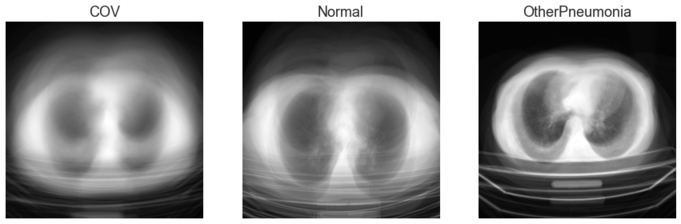
Przetwarzanie obrazów
Obrazy ze skanera CT zawierają szum, który nie jest istotny przy klasyfikacji, a wręcz może pogarszać wynik. Jak zobaczyliśmy na przykładowych obrazach takimi niepożądanymi elementami są tło oraz stół, na którym leży badany pacjent. W przypadku klasyfikacji na podstawie zmian w płucach właściwie wszystko, co nie jest płucami, należałoby usunąć z obrazu.
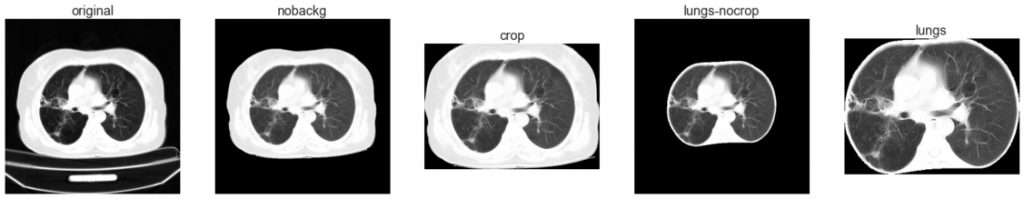
Wstępne przetwarzanie obrazów w tym projekcie obejmuje znalezienie konturów ciała pacjenta, usunięcie zaszumionego tła oraz stołu z obrazu, a następnie znalezienie na obrazie płuc. Przed podaniem do modelu rozmiar zdjęć jest ujednolicany, a wartości pikseli są normalizowane do zakresu 0-1.
W celu sprawdzenia, jak wstępne przetwarzanie obrazów wpływa na wyniki, modele trenowano na: obrazach oryginalnych (512 x 512 px), obrazach z wyciętym tłem i stołem (512 x 512 px), obrazach przyciętych do pacjenta (512 x 328 px), obrazach z wysegmentowanymi płucami (512 x 512 px) oraz obrazach przyciętych do płuc (330 x 256 px).
Usunięcie szumu
Do usunięcia szumów z obrazu została wykorzystana biblioteka OpenCV.
Pierwszym krokiem jest zbinaryzowanie obrazu:
retval, img_thresh = cv2.threshold(img, thresh=150, maxval=255, type=cv2.THRESH_BINARY)
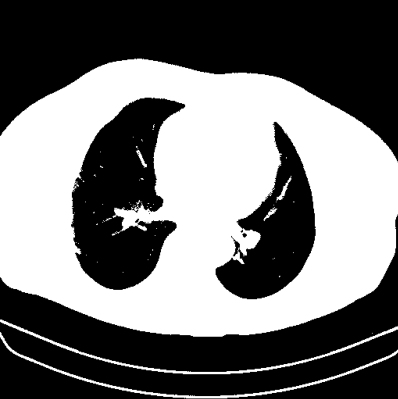
Do zbinaryzowanego obrazu dodawana jest ramka (ułatwi znalezienie największego obiektu – pacjenta – w przypadku, gdy pacjent będzie znajdował się przy krawędzi obrazu), kolory są odwracane, a następnie są znajdywane krawędzie:
img_border = cv2.copyMakeBorder(img_thresh, top, bottom, left, right, cv2.BORDER_CONSTANT, None, 0)
img_border_a = cv2.bitwise_not(img_border)
img_edges = cv2.Canny(img_border_a, 240, 255)
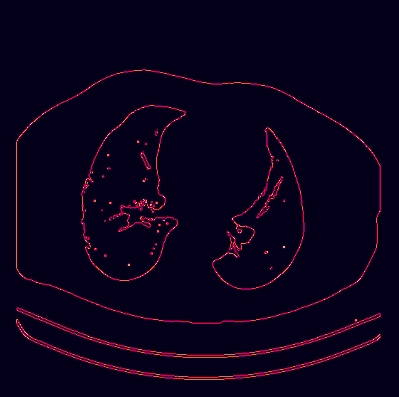
Aby domknąć krawędzie, robimy dylatację, po czym znajdujemy najdłuższy kontur:
kernel = np.ones((3, 3), np.uint8)
img_dilated = cv2.dilate(img_edges, kernel, 1)
contours, hierarchy = cv2.findContours(img_dilated, cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_SIMPLE)
max_index, max_area = max(enumerate([cv2.arcLength(contour, True) for contour in contours]), key=lambda x: x[1])
max_contour = contours[max_index]
max_contour = max_contour - top
img_body_contour = cv2.drawContours(img.copy(), [max_contour], 0, (0, 255, 0), 2)
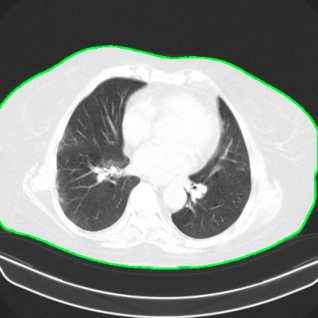
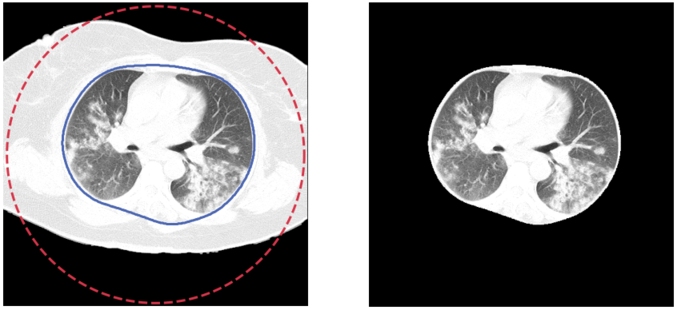
Na podstawie znalezionego konturu, tworzymy maskę i zostawiamy na obrazie tylko pacjenta:
mask = np.zeros((img.shape[0], img.shape[1]), dtype=np.uint8)
mask = cv2.fillPoly(mask, [max_contour], (1.0, 1.0, 1.0))
img_masked = cv2.bitwise_and(img, img, mask=mask)
Usunięcie szumu – napotkane problemy
Niestety przedstawione kroki nie są wystarczające w każdym przypadku. Zdarzają się sytuacje, w których najdłuższy kontur obejmuje również stół lub najdłuższy kontur ma płuco albo stół:
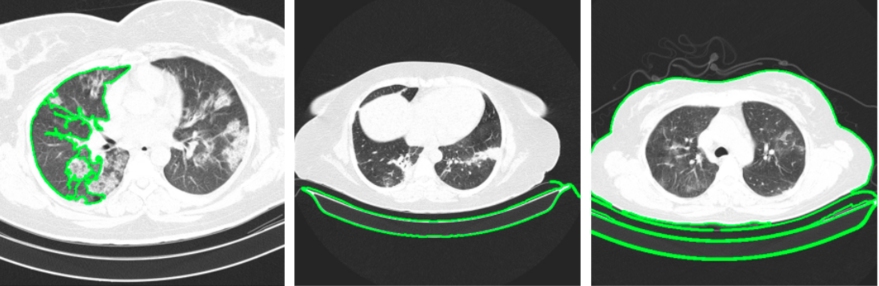
1. W przypadku, gdy najdłuższy kontur ma płuco, rozwiązaniem jest sprawdzenie, czy kontur wypukły (convex hull) jest dłuższy niż zwykły kontur:
if_contour = 1
max_contour = contours[0]
max_hull = cv2.convexHull(contours[0])
max_hull_perimeter = cv2.arcLength(max_hull, True)
for contour in contours:
hull = cv2.convexHull(contour)
if cv2.arcLength(contour, True) > cv2.arcLength(max_contour, True):
max_contour = contour
if_contour = 1
elif cv2.arcLength(hull, True) > max_hull_perimeter:
max_hull_perimeter = cv2.arcLength(hull, True)
max_hull = hull
max_contour = contour
if_contour = 0
2. Z kolei pozbycie się stołu można uzyskać przez zastosowanie erozji na zbinaryzowanym obrazie z ramką:
kernel = np.ones((3, 3), np.uint8)
img_border_a = cv2.erode(img_border, kernel, 10)
Poprawione kontury wyglądają teraz odpowiednio:
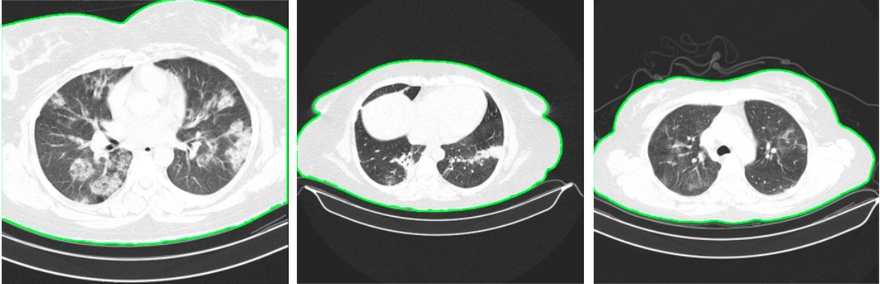
3. Innym napotkanym problemem było to, że czasem pacjent znajdował się zbyt blisko górnej krawędzi, co powodowało, że płuca nie znajdowały się wewnątrz znalezionego konturu:
Udało się to naprawić, poprawiając maskę poprzez jej „zamknięcie” przy górnej krawędzi i użycie floodFill:
non_zero_indices = np.argwhere(mask[0,:])
if non_zero_indices.size:
mask[0:5,int(non_zero_indices[0]):int(non_zero_indices[-1])] = 1
mask_border = cv2.copyMakeBorder(mask, top, bottom, left, right, cv2.BORDER_CONSTANT, None, 0)
mask_fill = np.zeros((mask_border.shape[0]+2, mask_border.shape[1]+2), dtype=np.uint8)
cv2.floodFill(mask_border, mask_fill, (0,0), 255)
mask_fill = cv2.bitwise_not(mask_fill)
mask = mask_fill[top:mask_fill.shape[0]-bottom-2, left:mask_fill.shape[1]-right-2]
mask = mask - np.min(mask)
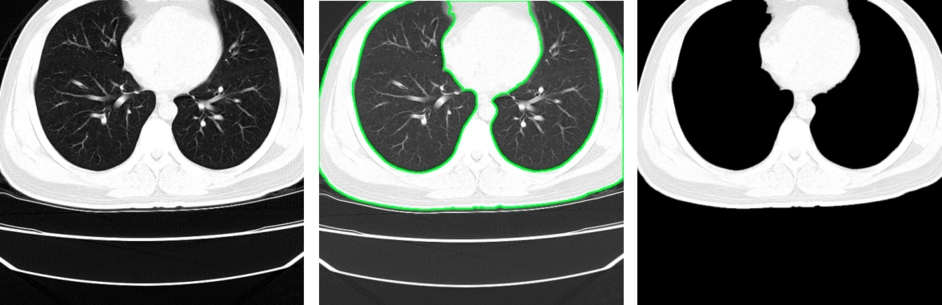
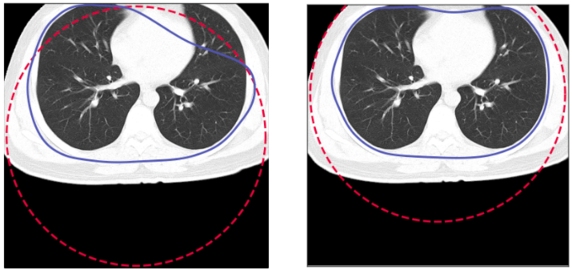
Segmentacja płuc
Kolejnym etapem przetwarzania wstępnego obrazów jest znalezienie płuc na obrazie przy użyciu modułu segmentation biblioteki scikit-image. Ponieważ mamy już przygotowane obrazy bez tła, wykorzystamy je w tym kroku.
Płuca znajdujemy, wykorzystując funkcję active_contour, która dopasowywuje początkowy kształt (snake) – tutaj okrąg – do cech obrazu. Na podstawie znalezionego konturu tworzymy maskę i zostawiamy na obrazie jedynie płuca.
def snake_contour(path, num_points=500, radius=250, alpha=0.1, beta=10, gamma=0.001, max_iterations=2500, w_line=1, w_edge=1, center=False, title=None, show_steps=False):
img = cv2.imread(path, cv2.IMREAD_GRAYSCALE)
non_zero_h = np.argwhere(img[:,int(img.shape[0]//2)])
center_h = (non_zero_h[0] + non_zero_h[-1]) // 2
non_zero_w = np.argwhere(img[int(img.shape[1]//2),:])
center_w = (non_zero_w[0] + non_zero_w[-1]) // 2
img = img_as_float(img)
s = np.linspace(0, 2*np.pi, num_points)
r = center_h + radius*np.sin(s)
c = center_w + radius*np.cos(s)
init = np.array([r, c]).T
snake = active_contour(gaussian(img, 3), init, alpha=alpha, beta=beta, gamma=gamma, coordinates='rc', max_iterations=max_iterations, w_line=w_line, w_edge=w_edge)
snake = np.asarray(snake, dtype=np.int32)
snake_contour = np.dstack((snake[:,1], snake[:,0]))
mask = np.zeros((img.shape[0], img.shape[1]), dtype=np.uint8)
mask = cv2.fillPoly(mask, [snake_contour], (1.0, 1.0, 1.0))
img_masked = cv2.bitwise_and(img, img, mask=mask)
return img_masked * 255
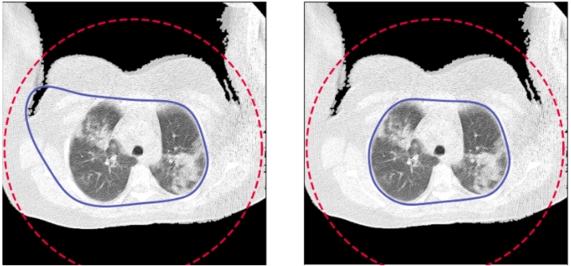
Segmentacja płuc – napotkane problemy
Zdarzyły się przypadki, w których snake nie od razu poradził sobie ze znalezieniem właściwego obszaru.
1. Gdy pacjent znajdował się zbyt blisko krawędzi, należało przesunąć początkowy okrąg tak, aby jego środek znajdował się na środku pacjenta:
2. Innym problemem były ręce znajdujące się na obrazie. W takim przypadku pomogło zwiększenie parametru alpha funkcji active_contour z 0.1 na 0.3:
3. W niektórych przypadkach okazało się, że początkowa liczba punktów w okręgu jest za mała, aby objąć oba płuca, dlatego została zwiększona z 400 na 500.
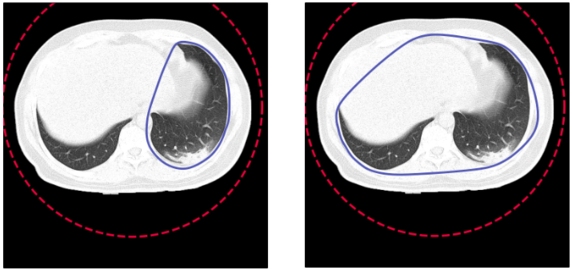
Niestety nadal może się zdarzyć, że kontur, poza płucami, otoczy również inne struktury tak jak w powyższym przykładzie lub inne, na przykład pętlę jelita. Jest to związane z wybraną metodą przetwarzania obrazów. Aby tego uniknąć, można by było zastosować model do segmentacji płuc, który powinien precyzyjniej poradzić sobie z tym zadaniem.
Data augmentation
Dla każdej grupy obrazów wymienionych na początku sekcji Przetwarzanie obrazów zastosowano te same techniki data augmentation: losowe odbicie poziome, losowy obrót, losowe przesunięcie oraz losowe przybliżenie, jednak w różnym zakresie. Przykładowo dla obrazów oryginalnych, na których płuca znajdują się na środku i stosunkowo daleko od krawędzi można zastosować większe przesunięcie niż w przypadku obrazów przyciętych do płuc.
| Obrazy | Horizontal Flip | Rotation | Translation | Zoom |
|---|---|---|---|---|
| Oryginalne | ✓ | 0.1 | 0.1 | -0.2 |
| Z usuniętym tłem | ✓ | 0.1 | 0.1 | -0.2 |
| Z usuniętym tłem – przycięte | ✓ | 0.07 | 0.07 | -0.1 |
| Płuca z usuniętym tłem | ✓ | 0.1 | 0.15 | -0.2 |
| Płuca z usuniętym tłem – przycięte | ✓ | 0.05 | 0.05 | -0.1 |
W zależności od modelu data augmentation uzyskano poprzez dodanie do modelu warstwy data augmentatio składającej się z warstw tf.keras.layers.experimental.preprocessing:
data_augmentation = tf.keras.Sequential([layers.experimental.preprocessing.RandomFlip('horizontal', seed=seed, input_shape=(img_height, img_width, num_channels)),
layers.experimental.preprocessing.RandomRotation(rotation, seed=seed, fill_mode='constant'),
layers.experimental.preprocessing.RandomZoom(zoom, seed=seed, fill_mode='constant'),
layers.experimental.preprocessing.RandomTranslation(translation, translation, seed=seed, fill_mode='constant')])
model = Sequential([data_augmentation,
model_base])
lub używając ImageDataGenerator z Kerasa:
datagen_train = tf.keras.preprocessing.image.ImageDataGenerator(rescale=1./255,
horizontal_flip=True,
rotation_range=rotation,
width_shift_range=translation,
height_shift_range=translation,
fill_mode='constant',
zoom_range=zoom)
train_generator = datagen_train.flow_from_directory(PATH_TRAIN,
classes=class_names,
color_mode=color_mode,
target_size=(img_height, img_width),
seed=seed,
batch_size=batch_size,
class_mode='categorical')
Modele
W ramach projektu sprawdzono kilka architektur. Część modeli była trenowana od zera, a w niektórych przypadkach zastosowano fine-tuning jedynie warstwy klasyfikującej. Jak wspomniano powyżej skuteczność modeli sprawdzono na pięciu rodzajach obrazów oraz z i bez data augmentation. Sprawdzono również, jak na wyniki wpływa ważenie klas (ang. class weight) – jest to jeden ze sposobów radzenia sobie z niezbalansowanym zbiorem danych.
Sprawdzone konfiguracje obejmują (w nawiasach są podane oznaczenia używane w dalszej części przy prezentacji wyników):
Modele:
- Simple – prosty model
- Tiny
- Small
- LargeW
- LargeT
- ResNet-50
- EfficientNet B3 pretrenowany na ImageNet-1k
- EfficientNet B3 trenowany od zera
Obrazy:
- oryginalne (original)
- z usuniętym tłem (nobackg)
- z usuniętym tłem, przycięte do pacjenta (crop)
- z wysegmentowanymi płucami (lungs-nocrop)
- przycięte do wysegmentowanych płuc (lungs)
Inne parametry i techniki:
- data augmentation (dataaug)
- class weight (classw)
- brak powyższych (baseline)
Prosty model
Dla sprawdzenia, jak bardzo poprawiają (lub pogarszają) wyniki zaawansowane architektury wytrenowany został również bardzo prosty model:
model = Sequential([normalization_layer,
Conv2D(16, 3, activation='relu', input_shape=(img_height, img_width, num_channels)),
MaxPooling2D(),
Conv2D(32, 3, activation='relu'),
MaxPooling2D(),
Conv2D(64, 3, activation='relu'),
MaxPooling2D(),
GlobalMaxPooling2D(),
Dense(32, activation='relu'),
Dense(num_classes)])
Poniżej znajdują się wyniki dla każdej z konfiguracji pogrupowane według obrazów, na których dany model był trenowany. Dla ułatwienia porównań przerywaną linią zaznaczony jest f1_score = 0.65.
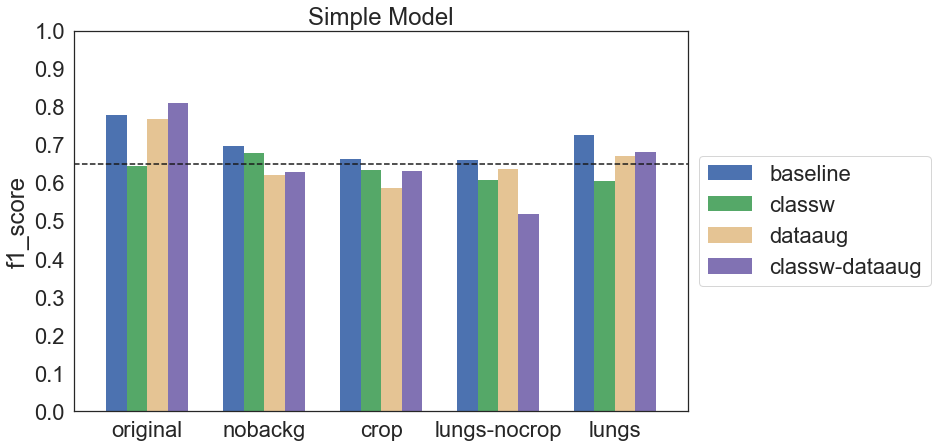
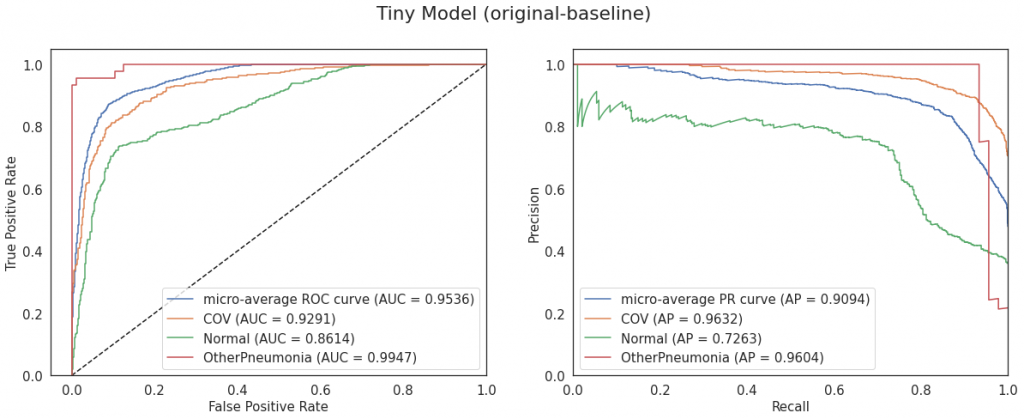
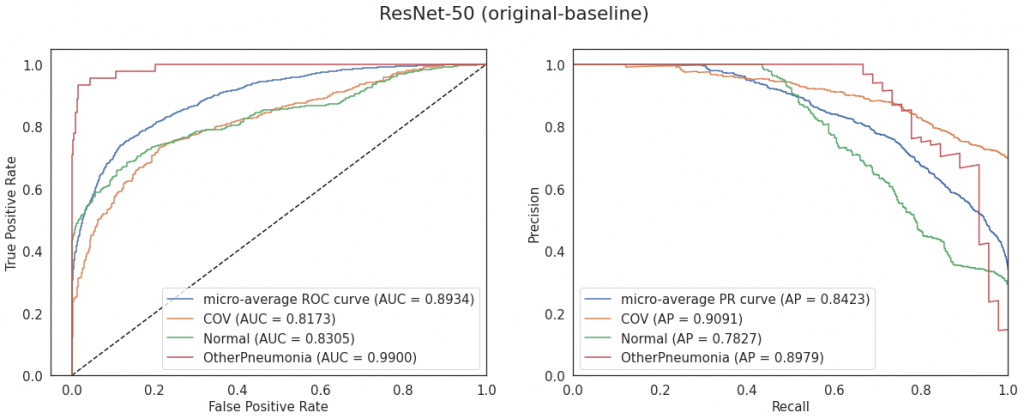
Okazuje się, że zastosowane techniki data augmentation niekoniecznie poprawiają wyniki, a mogą je wręcz pogorszyć. Podobnie jest z ważeniem klas. Zerknijmy na krzywe ROC i Precision-Recall modelu, który osiągnął najwyższy f1 score:
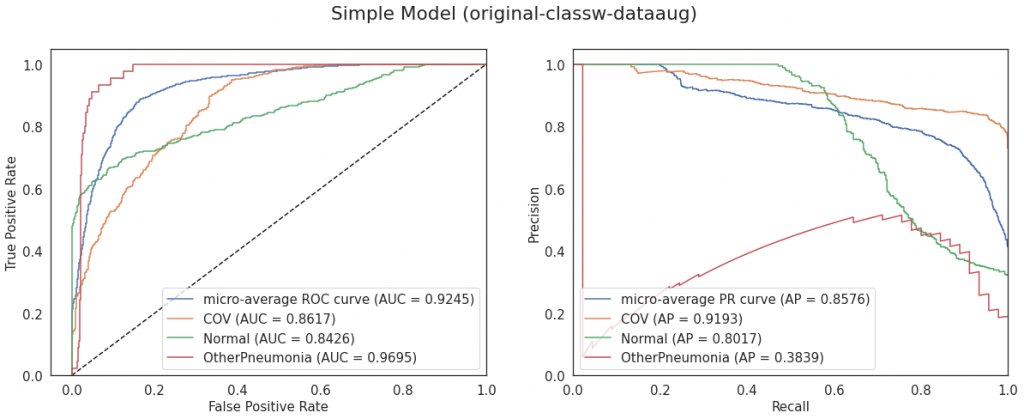
Prosty model ma dość wysokie AUC i Average Precision. Jedynie dla klasy OtherPneumonia Average Precision jest niskie; jest to klasa, dla której mamy najmniej przykładów w zbiorze, a jednocześnie może być podobna do COV. Zerknijmy jeszcze na znormalizowaną macierz pomyłek (normalizacja pomaga w interpretacji, szczególnie gdy mamy zbiór niezbalansowany):
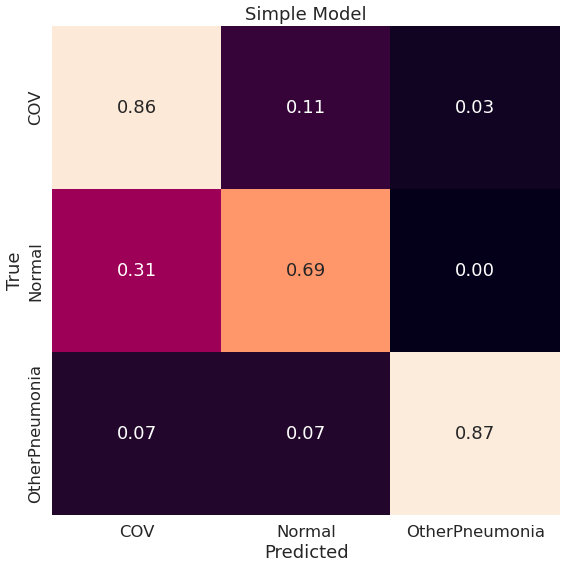
Model myli jeszcze trochę klasy COV i Normal, 31% klasy Normal zostało zaklasyfikowane jako COV. Jeśli chodzi o OtherPneumonia to widzimy, że większość przypadków zostało zaklasyfikowanych poprawnie, jednak musimy mieć na uwadze, że w zbiorze walidacyjnym jest ich jedynie 45, jak już zostało wspomniane powyżej.
Transfusion
Kolejnymi wykorzystanymi modelami są architektury Tiny, Small, LargeW, LargeT zaproponowane w artykule Transfusion: Understanding Transfer Learning for Medical Imaging. Modele były trenowane od zera.
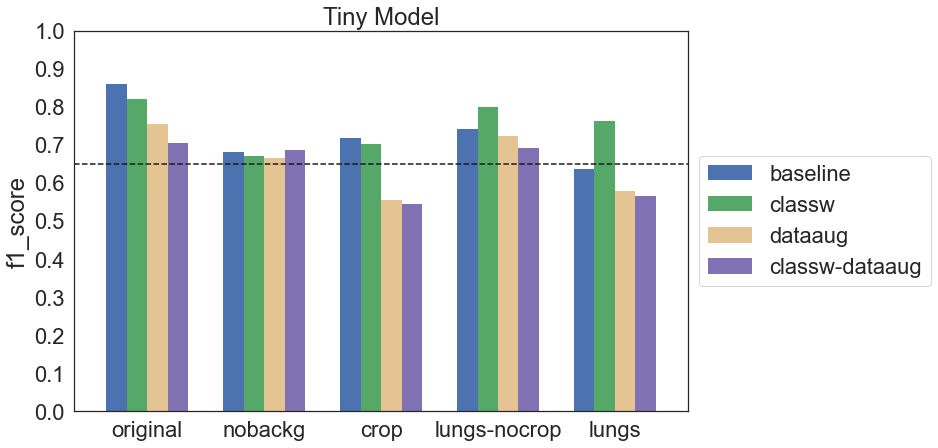
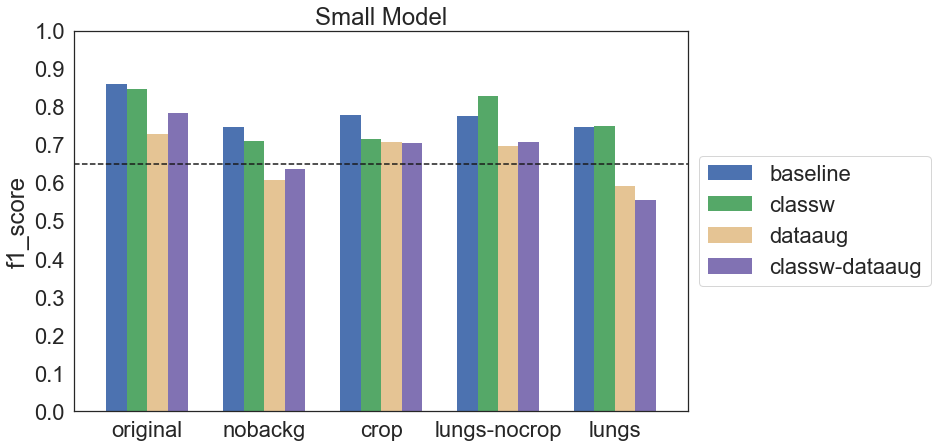
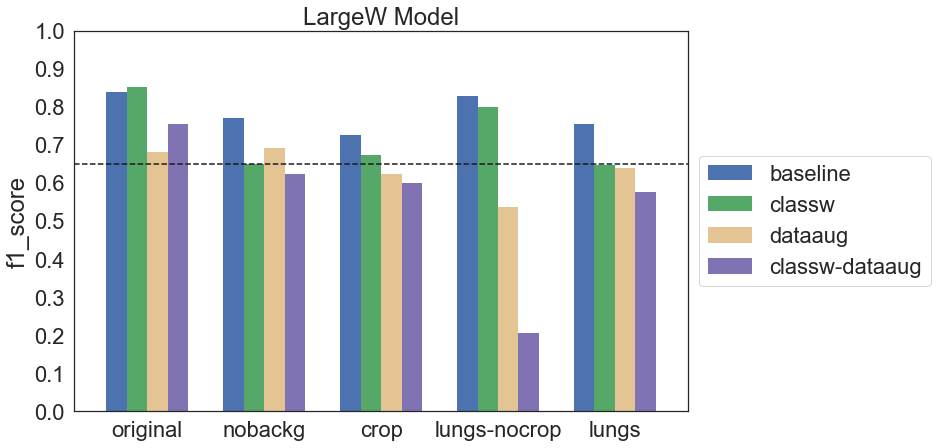
Podobnie jak w przypadku modelu Simple okazuje się, że zastosowanie data augmentation może pogarszać wyniki. W takim wypadku możliwe jest, że wybrane metody były nieodpowiednie dla danych obrazów lub użyte parametry były zbyt duże. Nieco lepszy efekt w tej grupie modeli miało ważenie klas – czasem poprawiało wynik, chociaż czasem różnica między baseline a classw była nieznaczna.
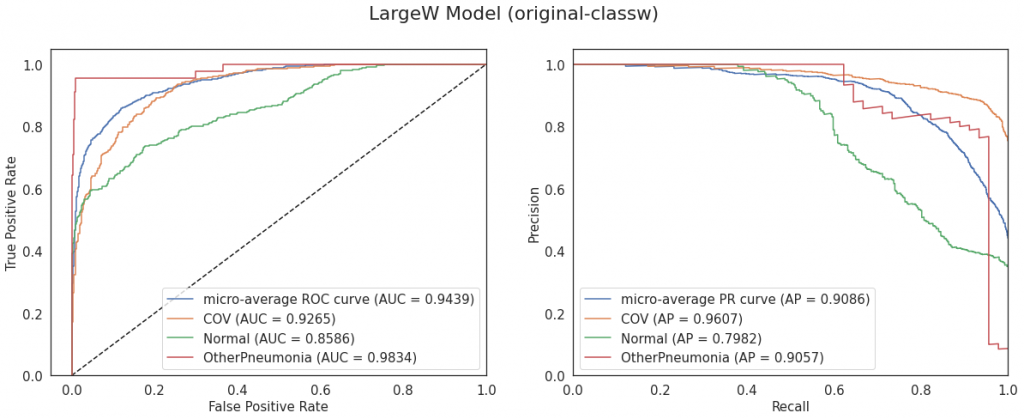
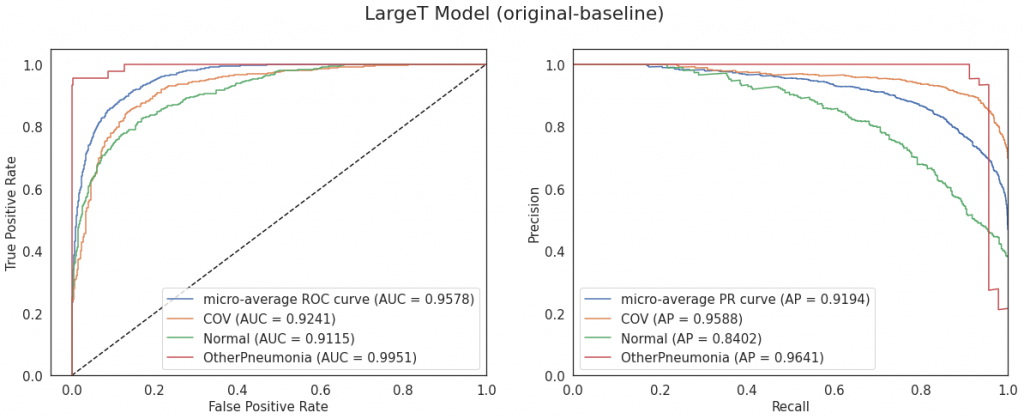
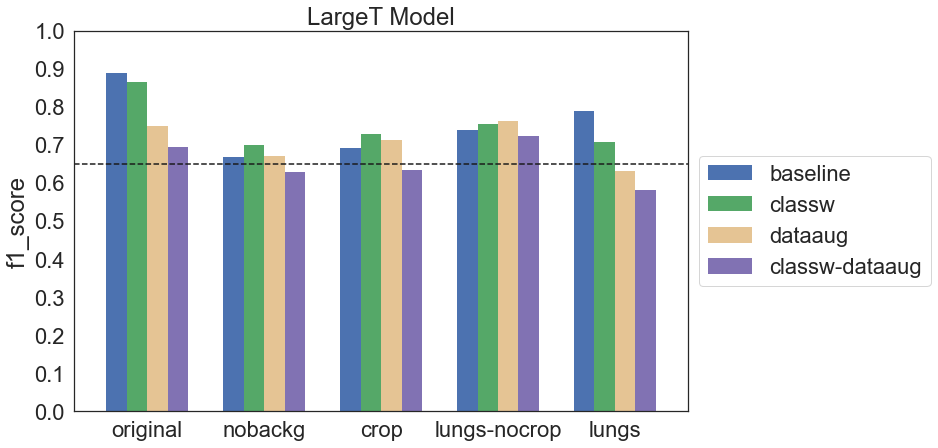
Najlepsze metryki z powyższych ma model LargeT, widać to również na poniższych macierzach pomyłek:
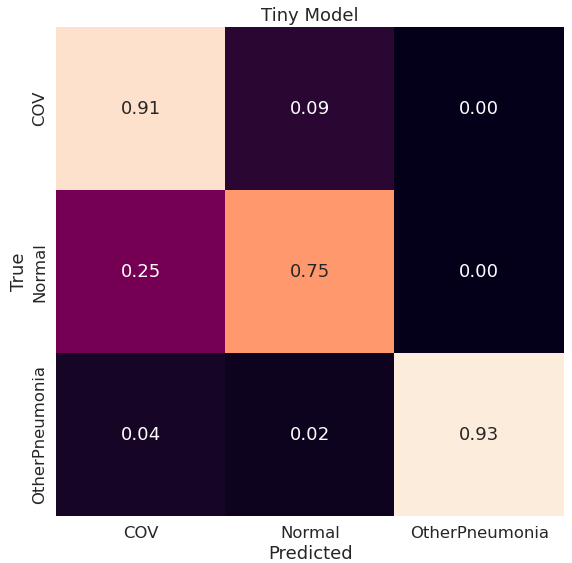
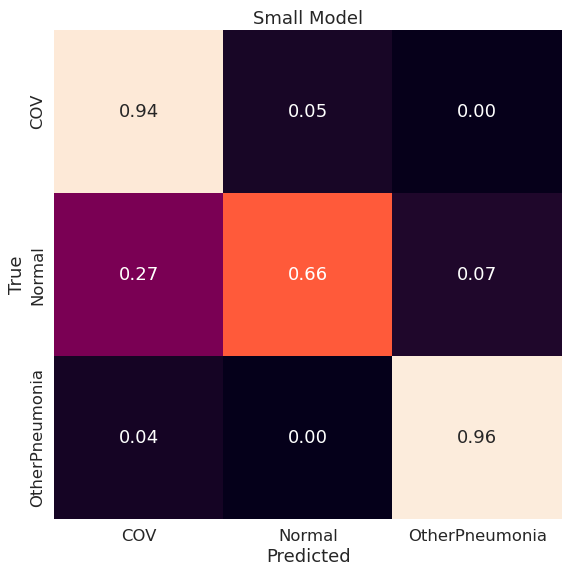
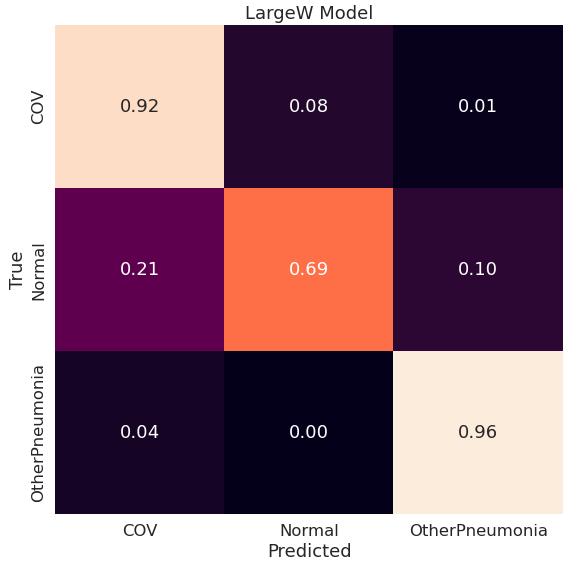
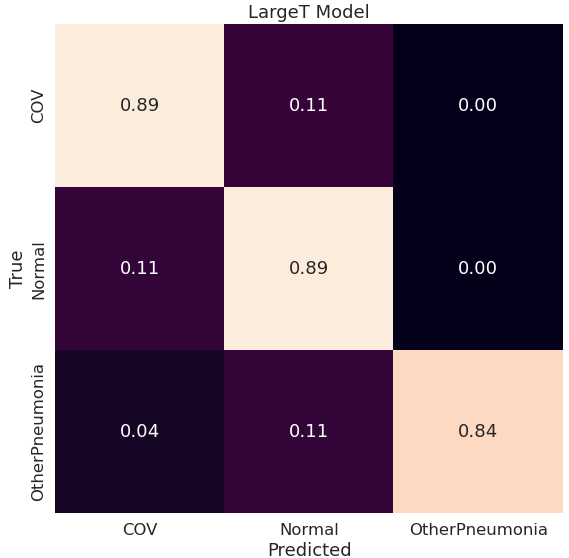
Google Big Transfer
Architektura zaczerpnięta z Google Big Transfer (A. Kolesnikov, L. Beyer, X. Zhai, J. Puigcerver, J. Yung, S. Gelly and N. Houlsby: Big Transfer (BiT): General Visual Representation Learning) to ResNet-50 (BiT-S R50x1). Model jest pretrenowany na ImageNet-1k i w tym projekcie dotrenowana została jedynie warstwa klasyfikująca.
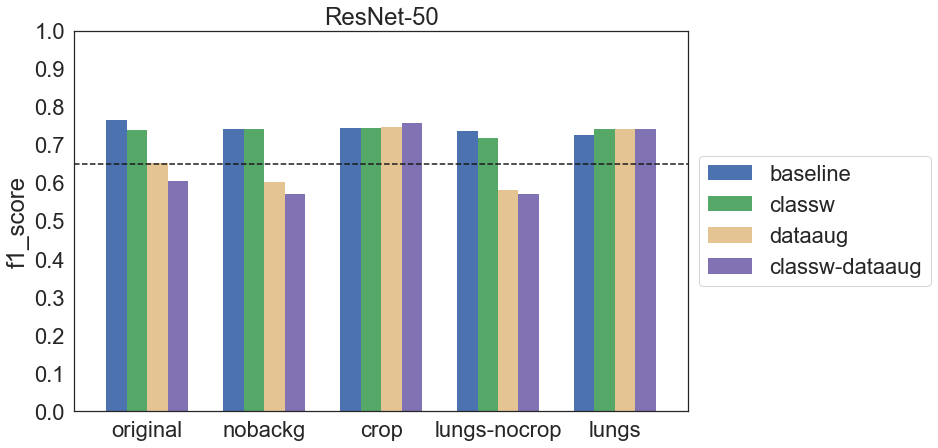
ResNet-50 dla obrazów przyciętych, na których praktycznie nie było tła (crop oraz lungs) osiąga równomierne wyniki. Z kolei w przypadku pozostałych obrazów ponownie okazuje się, że data augmentation pogarsza wynik.
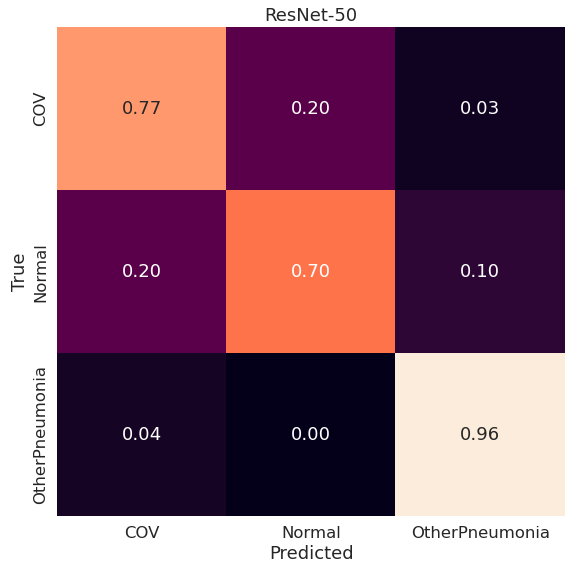
Podobnie jak w niektórych powyższych przypadkach część przypadków klasy Normal jest klasyfikowana jako COV, a COV jako Normal. OtherPneumonia jest klasyfikowane poprawnie w 96%.
EfficientNet B3
W przypadku EfficientNet B3 zastosowano dwa podejścia: trening całego modelu od początku oraz transfer learning. W drugim podejściu wykorzystano model pretrenowany na ImageNet dostępny w Keras Applications i dostrojono jedynie warstwę klasyfikującą.
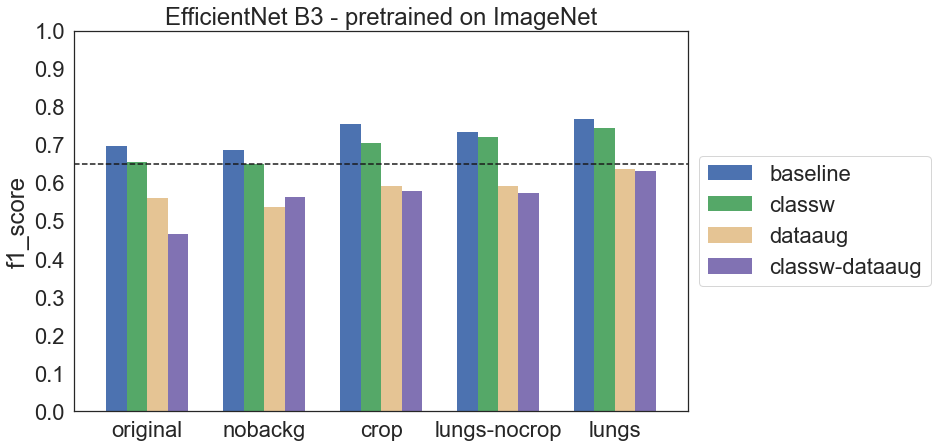
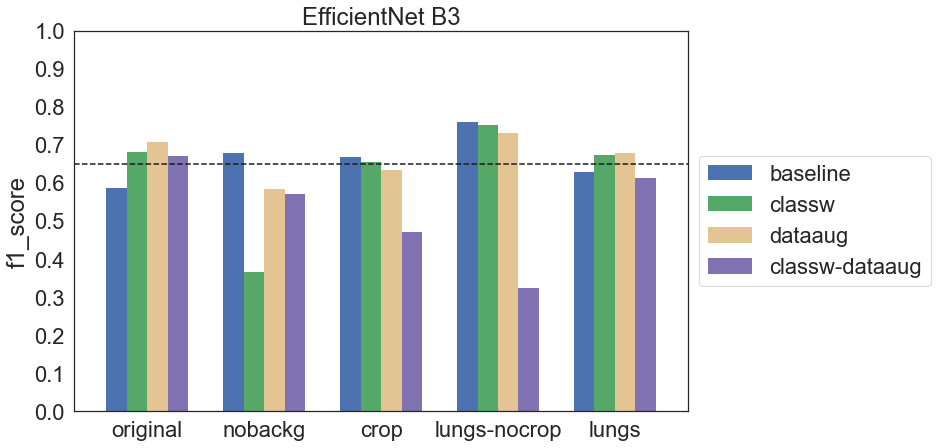
Widać pewne różnice pomiędzy wariantami EfficientNet B3. Są także pewne podobieństwa – w wielu przypadkach data augmentation wpłynęło niekorzystnie na wyniki. Również ważenie klas nie poprawiało znacząco wyników.
Uwzględniając konfiguracje bez data augmentation (które okazuje się pogarszać wyniki) można stwierdzić, że lepszym podejściem w przypadku tych danych jest transfer learning. Wpływa na to między innymi liczba obrazów – jest ich mało, co utrudnia skuteczne trenowanie dużych modeli.
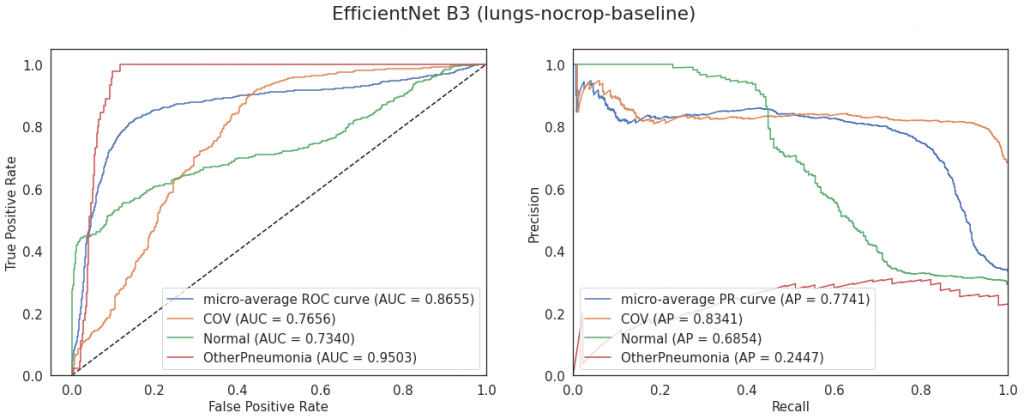
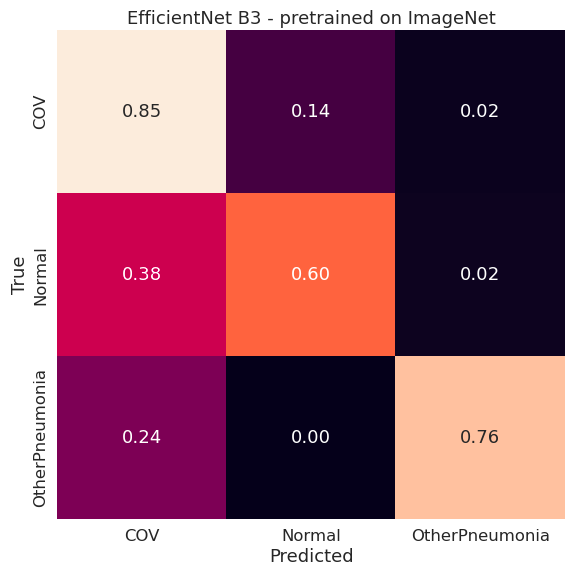
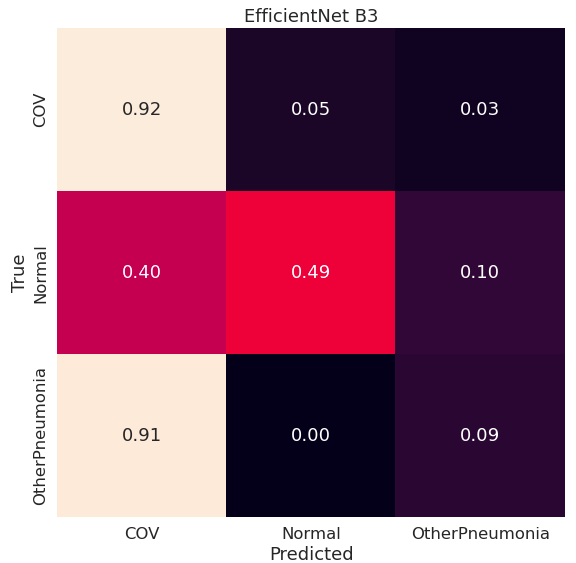
Porównanie modeli EfficientNet
Oprócz EfficientNet B3 na zdjęciach oryginalnych wytrenowane zostały jeszcze warianty B0 i B7 jako dodatkowy test sprawdzający, czy bardziej rozbudowany model poradzi sobie lepiej. Poniżej na wykresie znajdują się wyniki dla poszczególnych architektur.
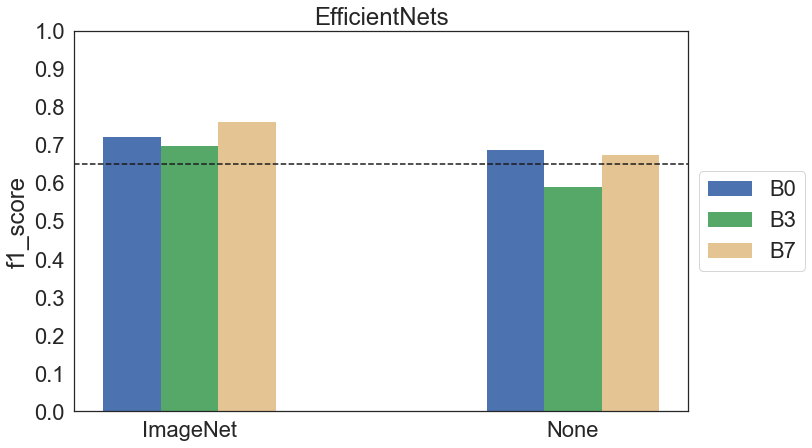
Widzimy, że – podobnie jak B3 – B0 oraz B7 trenowane od zera radzą sobie nieco gorzej niż wersje pretrenowane. W przypadku modeli pretrenowanych B7 osiągnął trochę lepszy wynik od pozostałych, z kolei w przypadku modeli trenowanych od zera wynik B7 różni się nieznacznie od B0. Wstępnie można stwierdzić, że faktycznie B7 daje dokładniejsze wyniki, przynajmniej dla transfer learningu, jednak do wysnucia pewniejszych wniosków należałoby przeprowadzić więcej eksperymentów.
Podsumowanie walidacji wszystkich modeli
Wszystkie modele trenowały się stabilnie, natomiast walidacja podczas treningu była dość niestabilna. Dla większych modeli (ResNet-50, EfficientNet B3) wahania metryk i lossu były znacznie mniejsze niż u innych modeli, natomiast największe wahania zaobserwowano u modeli Transfusion.
W przypadku wszystkich modeli data augmentation negatywnie wpływało na wyniki (oprócz ResNet-50 trenowanego na obrazach crop oraz lungs). Z kolei ważenie klas w niektórych przypadkach poprawiło wynik, w niektórych pogorszyło, a w jeszcze innych nie zmieniło znacząco.
Poniżej w tabeli znajduje się zestawienie najlepszych modeli z każdej grupy wraz z konfiguracją i metrykami. Najwyższe metryki osiągnął model LargeT na obrazach oryginalnych bez ważenia klas i data augmentation.
| Model | Konfiguracja | F1 Score | AUC |
|---|---|---|---|
| Simple | original-classw-dataaug | 0.810 | 0.925 |
| Transfusion Tiny | original-baseline | 0.861 | 0.954 |
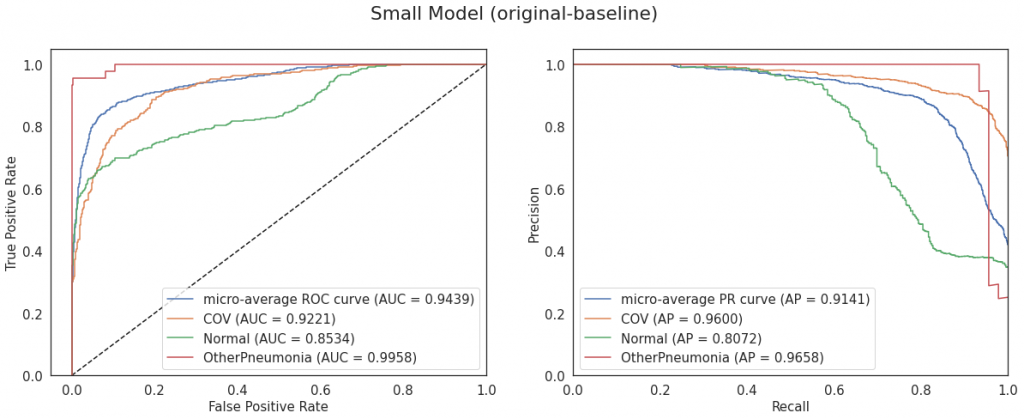
| Transfusion Small | original-baseline | 0.860 | 0.944 |
| Transfusion LargeW | original-classw | 0.853 | 0.944 |
| Transfusion LargeT | original-baseline | 0.888 | 0.958 |
| Google BiT ResNet-50 | original-baseline | 0.766 | 0.893 |
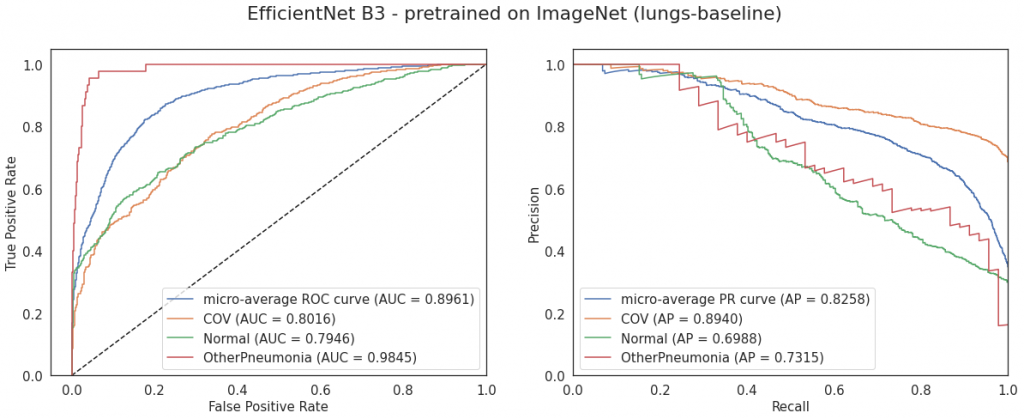
| EfficientNet B3 pretrained on ImageNet | lungs-baseline | 0.768 | 0.896 |
| EfficientNet B3 | lungs-nocrop-baseline | 0.761 | 0.866 |
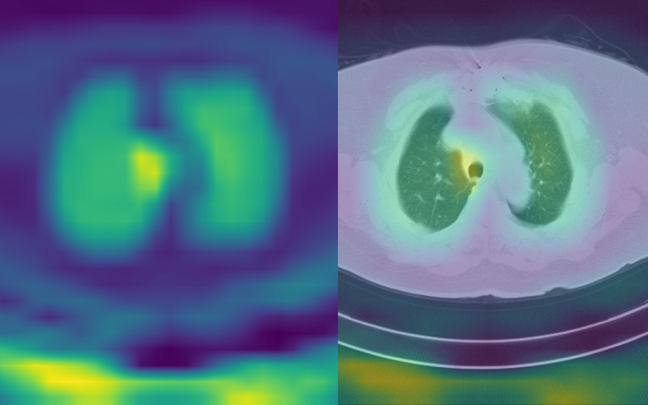
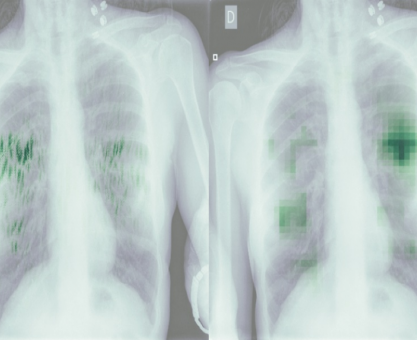
Gradient-weighted Class Activation Mapping
W medycynie istotne jest, aby dane rozwiązanie było interpretowalne, na przykład dodatkową odpowiedzią modelu może być heatmapa pokazująca, na jakie obszary obrazu model zwraca uwagę przy podejmowaniu decyzji. Może się okazać, że są to inne fragmenty niż te, na podstawie których decyzje podejmuje lekarz.
Jedną z metod interpretacji wyników jest Gradient-weighted Class Activation Mapping (Grad-CAM). Jest to generalizacja metody Class Activation Maps (CAM) i w porównaniu do niej nie wymaga konkretnej architektury modelu. Grad-CAM, podobnie jak CAM, wykorzystuje informacje z ostatniej warstwy konwolucyjnej (feature maps). Jednak wagi do obliczenia wynikowej heatmapy nie są brane z ostatniej warstwy fully connected, ale są liczone na podstawie gradientów.
Zobaczmy wyniki GradCAM dla najlepszego modelu według metryk – LargeT – dla poszczególnych grup obrazów (przykłady klasy COV):



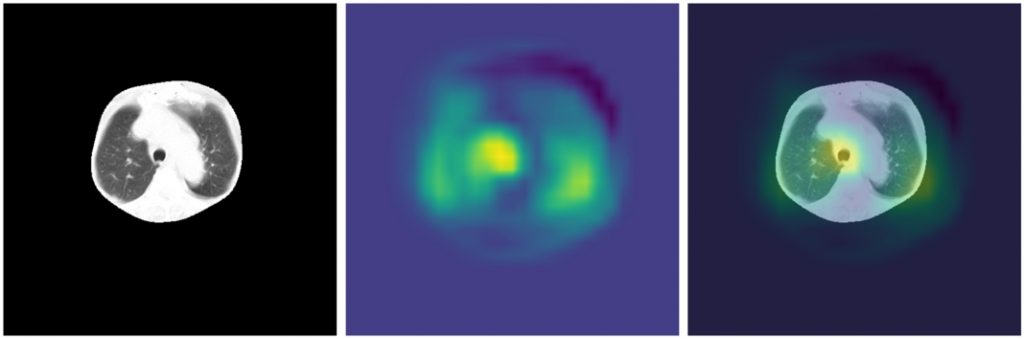
W pierwszym przykładzie widać, że model skupia się na płucach. Jednak poza tym zaznacza wyraźniej również tchawicę. Widać także, że istotne są dla modelu obszary wokół stołu, co potwierdza założenie, że stół należy usunąć z obrazu, gdyż nie wpływa on na diagnozę, a może wprowadzać model w błąd.
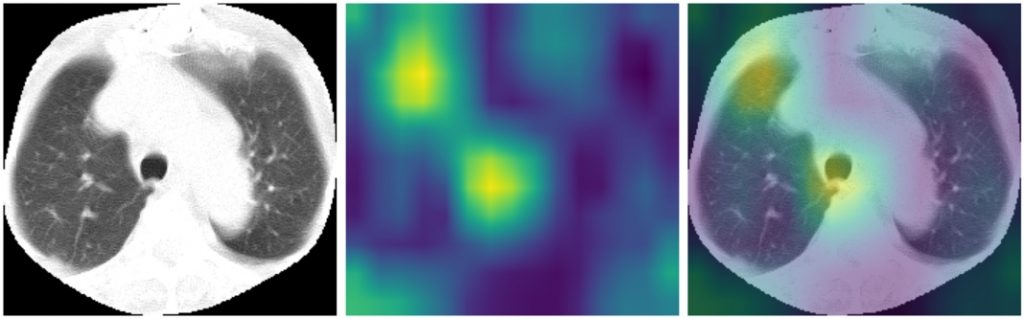
Dla jednego obrazu możemy wygenerować heatmapy odpowiadające każdej z klas. Poniżej znajdują się przykłady obrazu klasy COV oraz heatmapy uzyskane względem COV, Normal, OtherPneumonia (kolejno od góry):


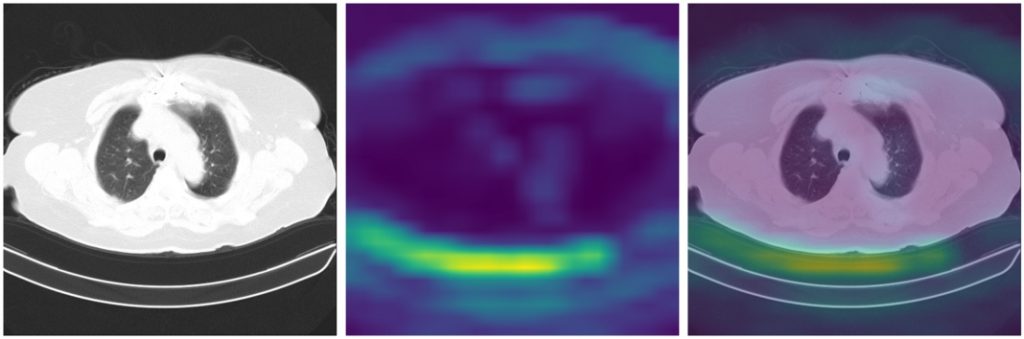
Widać, że reprezentacje z ostatniej warstwy konwolucyjnej są różne dla poszczególnych klas, przy czym w każdym przypadku jest zaznaczany niepożądany obszar wokół stołu.
Zerknijmy jeszcze na heatmapy dla poszczególnych klas wygenerowane na podstawie samych płuc, tym razem dla modelu EfficientNet B3 – ImageNet (lungs):
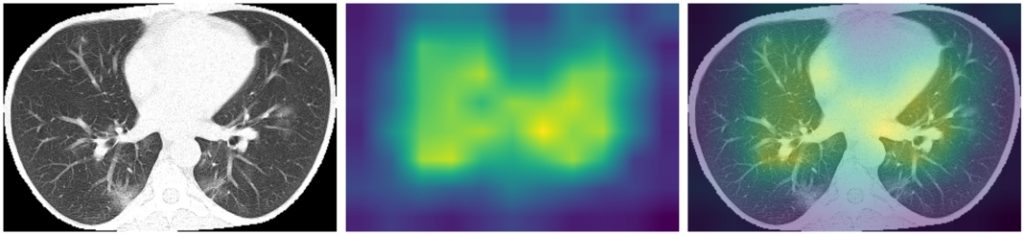
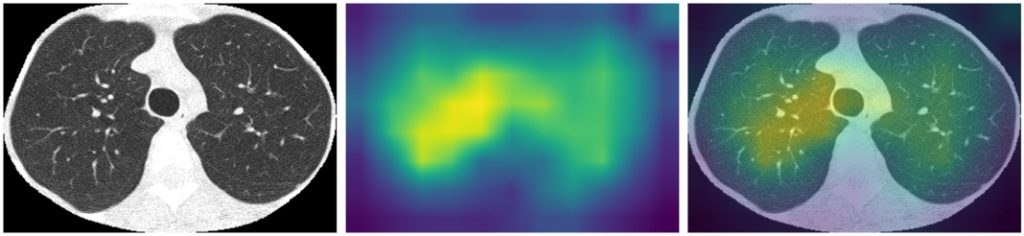
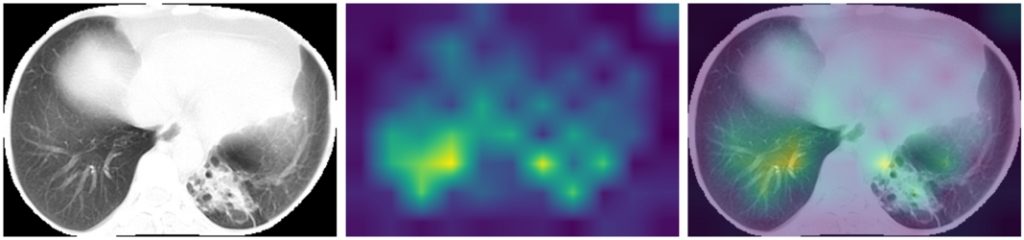
Widać, że dla modelu istotne są płuca, które na heatmapie można by nawet rozróżnić jako dwa. Jednak są one traktowanego raczej całościowo i model nie wyszukuje konkretnych zmian patologicznych.
Dla porównania spójrzmy jeszcze na heatmapy dla EfficientNet B3 trenowanego od zera:
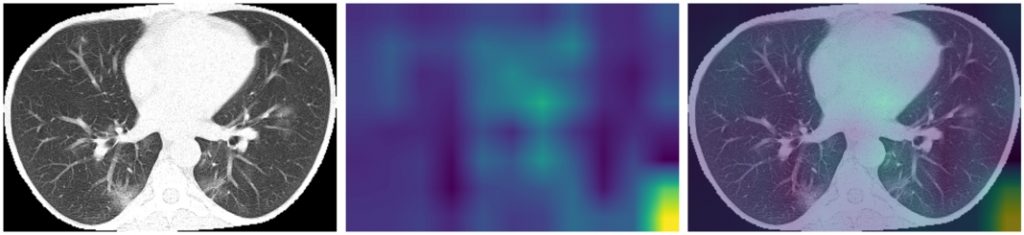
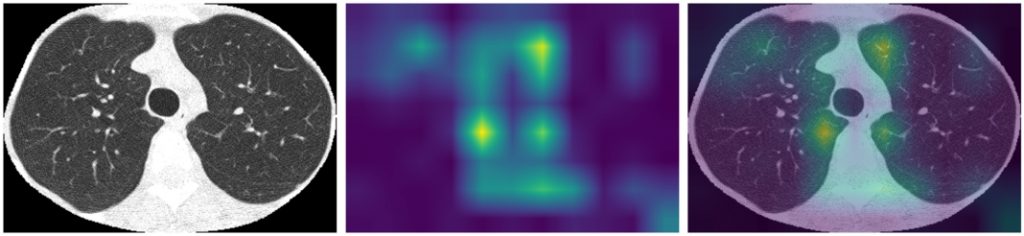

Widać, że model trenowany od zera wyłapuje inne cechy obrazu niż dostrojony model z wagami ImageNet, przy czym część istotnych dla modelu fragmentów obrazu jest nieistotna z medycznego punktu widzenia (na przykład obszary poza płucami). Może to wynikać ze zbyt małego zbioru danych – model nie jest w stanie nauczyć się ważnych cech na takim małym zbiorze treningowym.
Zbiór testowy
Ostatnim krokiem w projekcie jest przetestowanie modeli na zbiorze testowym. Okazuje się, że zbiór ten ma podobne proporcje jak zbiór treningowy i walidacyjny, tj. klasy COV jest najwięcej, a OtherPneumonia najmniej.
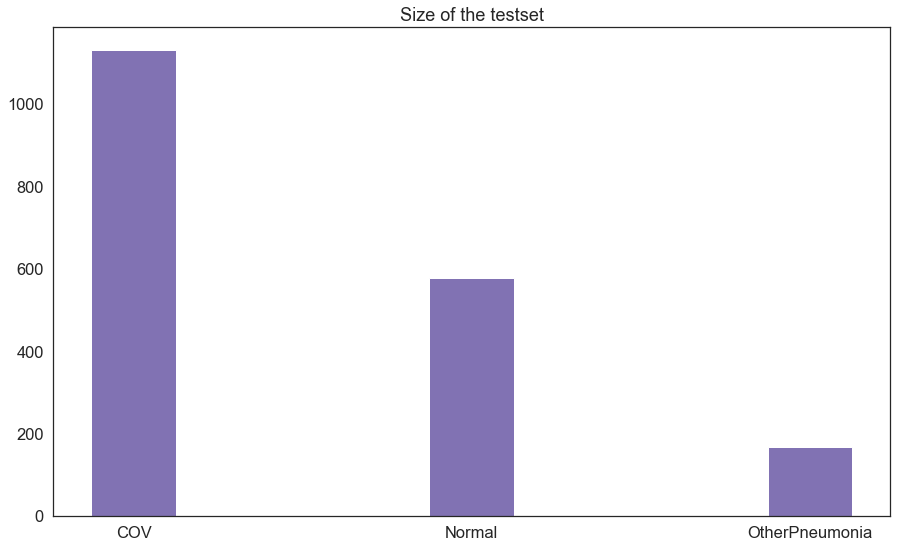
Sprawdzone zostały modele, które osiągnęły najwyższe wyniki na zbiorze walidacyjnym.
| Model | Konfiguracja | F1 Score | AUC |
|---|---|---|---|
| Simple | original-classw-dataaug | 0.582 | 0.810 |
| Transfusion Tiny | original-baseline | 0.580 | 0.811 |
| Transfusion Small | original-baseline | 0.600 | 0.798 |
| Transfusion LargeW | original-classw | 0.634 | 0.792 |
| Transfusion LargeT | original-baseline | 0.577 | 0.796 |
| Google BiT ResNet-50 | original-baseline | 0.711 | 0.886 |
| EfficientNet B3 pretrained on ImageNet | lungs-baseline | 0.629 | 0.845 |
| EfficientNet B3 | lungs-nocrop-baseline | 0.570 | 0.760 |
Wszystkie modele osiągnęły na zbiorze testowym niższe niż na zbiorze walidacyjnym wartości f1 score oraz AUC. Najwyższe wyniki na zbiorze testowym osiągnął ResNet-50. Wyniki te nie różnią się znacznie od metryk tego modelu dla zbioru walidacyjnego, a więc ResNet-50 okazał się modelem o najlepszej generalizacji – najlepiej rozpoznaje nowe obrazy, spoza zbioru treningowego, a także walidacyjnego (do którego niejako dostrajaliśmy skuteczność modeli w trakcie ich rozwijania i poprawy wyników).
W tym miejscu warto by było przeprowadzić analizę gradCAM dla ResNet-50 i sprawdzić, dlaczego osiągnął lepsze wyniki niż pozostałe modele oraz czy podejmuje decyzje na podstawie właściwych cech. Niestety otrzymanie wyjścia z ostatniej warstwy konwolucyjnej okazało się problematyczne ze względu na format, w jakim są zapisane modele Google Big Transfer – TensorFlow SavedModel – a także ze względu na opakowanie zapisanego modelu w KerasLayer podobnie jak w przykładowym notebooku. Z tego powodu pomijamy tymczasem analizę gradCAM dla ResNet-50. Jeśli ktoś z czytelników ma pomysł na rozwiązanie tego problemu – sugestie mile widziane.
Podsumowanie
Analiza zbioru CT-COV19 okazała się niełatwym zadaniem, między innymi ze względu na niepodanie przez autora zbioru dokładnej charakterystyki obrazów. Brak informacji o zbiorze oraz/lub brak wiedzy domenowej może powodować, że podejmiemy błędne założenia w trakcie projektu, co doprowadzi do wadliwego rozwiązania. Dobrze jest konsultować założenia oraz wyniki z radiologami i innymi ekspertami. W tym kontekście warto zapoznać się z już wspomnianą wyżej checklistą, a także tym artykułem przeglądowym (autorzy artykułu konkludują, że żadne z rozwiązań machine learning powstałych w minionym roku do wykrywania i prognozowania COVID-19 na podstawie obrazów CT oraz RTG niestety nie nadaje się do użytku klinicznego). Oba opracowania listują błędy i nieścisłości rozwiązań uczenia maszynowego dla wykrywania i przewidywania COVID-19 oraz zawierają wytyczne, jak tych błędów unikać.
Preprocessing obrazów i ostatecznie segmentacja płuc nie przyniosły oczekiwanej poprawy wyników. Oprócz modelu EfficientNet B3 pozostałe modele podczas walidacji osiągnęły najwyższe wyniki na obrazach oryginalnych. Ten rezultat może być przypadkowy w tym kontekście, że walidacja podczas treningu była niestabilna, a checkpoint danego modelu był zapisywany na podstawie najwyższego f1 score walidacji po każdej epoce. W tej sytuacji na pewno mogłaby pomóc większa liczba obrazów w zbiorze zarówno treningowym, jak i walidacyjnym. Zwłaszcza, że analiza GradCAM pokazuje, że modele mogą podejmować decyzje na podstawie cech nieistotnych z medycznego punktu widzenia. Prawdopodobnie zbiór jest po prostu za mały, aby nauczyły się istotnych cech.
Ponieważ zbiór jest dość mały jak na wymagania sieci neuronowych, całkiem dobrze sprawdził się transfer learning (co widać m.in. na heatmapach gradCAM – model pretrenowany daje sensowniejsze wyniki w porównaniu do niepretrenowanego). Ogólnodostępne gotowe do użycia modele są najczęściej wytrenowane na zbiorze ImageNet. Pewnym problemem jest to, że zbiór ten zawiera jedynie obrazy naturalne (czyli na przykład zwierzęta, przedmioty codziennego użytku), które znacząco różnią się od obrazów medycznych. Najkorzystniejszym rozwiązaniem byłoby wykorzystanie modelu wytrenowanego na dużym zbiorze obrazów medycznych pochodzących z konkretnej modalności i dostrojenie go na posiadanym małym zbiorze. Niestety takie modele nie są powszechne dostępne (podobnie jak duże zbiory obrazów medycznych) i jeśli posiadamy mały zbiór nadal zasadne może być wykorzystanie modelu pretrenowanego na ImageNet (ciekawym podejściem do zoptymalizowania transferu wiedzy między obrazami naturalnymi a medycznymi jest pokolorowanie obrazów medycznych).
W jakim kierunku można dalej rozwijać projekt oraz jak poprawić wyniki? Przede wszystkim dobrze by było zdobyć większą ilość danych. Jak już było wspomniane obrazów jest dość mało, aby modele mogły nauczyć się poprawnie istotnych cech. Można by również wytrenować model na wszystkich obrazach naraz (oryginalnych, przyciętych, z usuniętym tłem). Kolejnym krokiem mogłoby być wypróbowanie innych technik data augmentation, np. zmiana kontrastu, CLAHE, czy zastosowanie filtru Frangiego.